生物は地球上のあらゆる環境で生息しており、強酸、強アルカリの環境や温泉 などの高温、さらには海底の高圧下の条件下でも多くの生物を見ることができる。 このような極端条件下で生きる生物もわれわれと同じように遺伝子をもち、蛋白質 から成り立っており、多くの生物からその機能は同じであるが耐酸性や耐熱性など の外的条件への応答の違った酵素が単離されている。これらの事実から、生物の構 成情報単位である遺伝子産物の蛋白質に特別な工夫がなされていると考えられてお り、その立体構造の研究は生物の機能解明のために重要であると考えられる。
高度好熱菌であるThermus thermophilus HB8は大島泰郎博士(現在、東 京薬科大学教授)らによって伊豆の温泉で発見、単離された耐熱性菌で多くの研究 の対象となっている。そのうち、NAD+を補酵素としロイシン生合成に関与する 3-isopropylmalate dehydrogenase(以後、IPMDHと略する)についてその遺伝 子の解析および大量発現、耐熱性についての研究が活発になされ、いくつかのアミ ノ酸の変異によってその熱的特性が大きく変化することが報告されている。また、 IPMDHについては耐熱性の違う細菌から抽出、精製されており、その熱的特性と 一次構造との関連について研究されている。したがって、本研究では、酵素の耐熱 性の機構を構造化学的に明らかにするため高度好熱菌Thermus thermophilsの 産するIPMDH、蛋白質工学の手法によって遺伝子融合によって作製された常温菌 であるBacillus subtilisの産するIPMDHとのキメラ酵素、および、それらの種々 の耐熱性を持った変異体の構造を決定してきた(表1)。

酵素の耐熱性の尺度の定義は非常に難しいが、残存活性が半分になる温度 (t1/2)でその酵素の耐熱性を評価できる。すなわち、酵素液を種々温度に保ち、一 定時間毎に液を取り出し活性測定の所定温度まで冷却の後にその酵素活性を測定す ればその変化率を時間についてプロットすればアレニウス・プロットでその勾配よ り反応のエネルギーを切片より活性を見ることができる。すこし簡便な方法として は温度は変化させるが測定は一定時間においてのみ行うことでも相対的な活性を知 ることができる。表1にはこのようにして測定したt1/2の値も示してある。耐熱性 の測定はCDスペクトルの温度変化やDSCによっても行われているが、基本的に は残存活性の測定と同じ結果を与えている。
1 これまでの解析結果
1-1 T.thermophilusの産するIPMDH
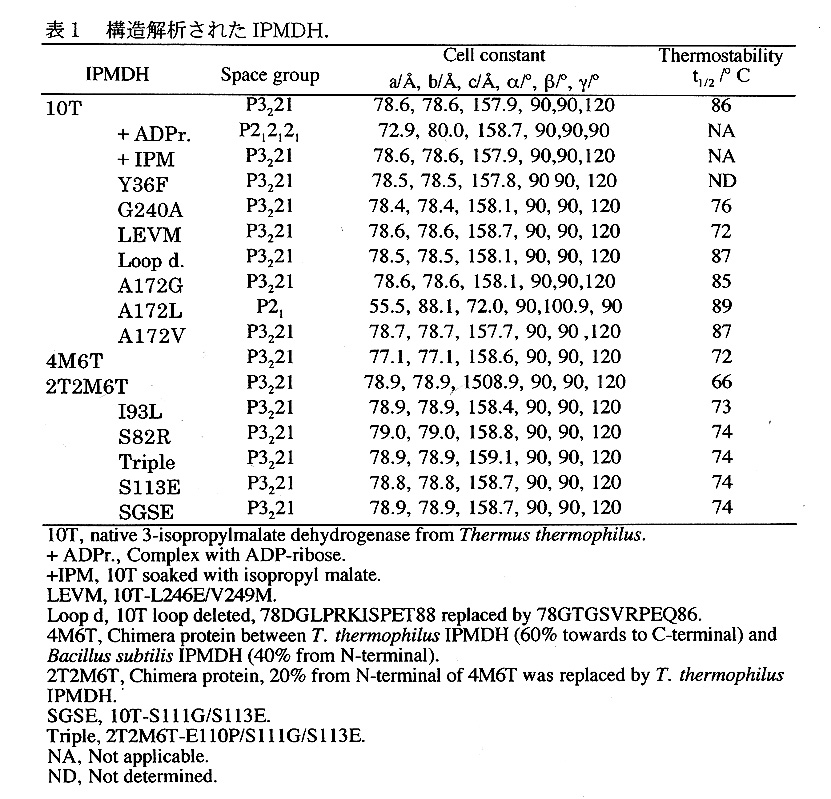
IPMDHは345残基のアミノ酸から成るサブユニットが二つ集まって分子を構 成している。本結晶では非対称単位がひとつのサブユニットで構成されており、分 子は結晶学的2回軸によって関係づけられているサブユニットで構成されている。 したがって、ふたつのまったく同じサブユニットが分子を作っているが、サブユニ ット自身も残基1-99と245-345からなるドメインと100-245から なるドメインがL254付近のループを介して結合して成り立っている(図1)。
本酵素の主鎖の構造はα/βの構造から成り立っており、サブユニットの中心部は 11本のβ鎖からなるシートで構成されており、β一シートの周囲を8本のα-ヘ リックスが取り囲んで外壁を形作っている。主鎖の構造での大きな特長は、1本の β鎖が分子表面より突き出ており、もうひとつのサブユニットの同じβ鎖と水素結 合を作りダイマーの安定化に寄与している。ダイマーを安定化しているもう一つ の力は245番近辺の疎水相互作用であろう。
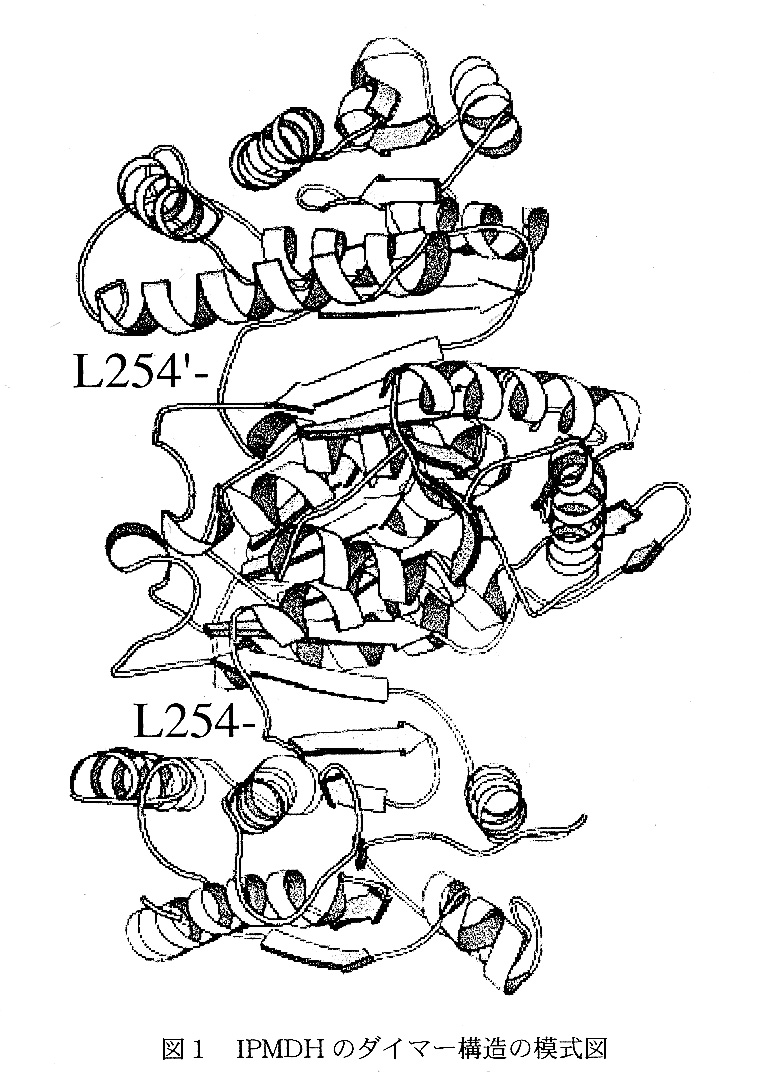
それでは本酵素の活性部位およびNAD+結合部位はどこであろうか。Zharang と Koshiland, Jr. は計算によってその構造を予測したが、我々は実験的にこれらの 部位を見出した。NAD+との複合体は共結晶化法によって作製したので空間群や非 対称単位中の分子数などが元の結晶と違っていた。結合位置は図2に示すように補 酵素や基質の結合を合理的に説明できるもので、前者では面の重なり合いと電荷の 中和、後者では金属が関与する結合状態が確認できた。
基質複合体は基質である3-isopropylmalateとMn2+を溶かした溶液に結晶を浸漬することによって作製に成功した。浸漬法を用いたので結晶は元の結晶と同形 で解析は容易に行うことができた。分子置換法によって得られた解に基づいたD- フーリエ図からMn2+の位置を容易に同定できMn2+と結合した基質も見出すことが できた。同族と考えられているisocitrate dehydrogenaseはIPMDHと同じ構造 の成り立ちをしておりサブユニットのドメイン間に活性部位があり基質と結合する とドメイン間が狭まると報告されている。IPMDHの場合、結晶ではそのような構 造変化は観測されなかったが、基質濃度などを高めると結晶が白濁し破壊されるこ となどから考えて、基質の結合により構造が変化することが期待できるので簡単な モデル作成の計算を行った。条件としてはサブユニットをふたつのドメインに分け 各々の回転、並進によってエネルギーが最小になるようした。その結果、IPMDH でもドメイン間の「閉じた構造」が可能であることを示すことができた。この事実 に基づいて、我々は「閉じた構造」のIPMDHの存在を期待して、種々の変異体の 構造の解析を行ったところ、後述のA172Lにおいて「閉じた構造」を見出したが、 耐熱性は向上したが酵素反応には目立った改善はみられなかった。
1-2キメラ酵素4M6T,2T2M6Tの構造
T.thermophilus由来の酵素(10T)とB.subtilis由来の酵素(10M)は種々の割 合で融合しその中間体であるキメラ酵素が作製されている。4M6TはN末端から4 割、すなわち、約130残基がB.subtilis由来で他がT.thermophilus由来であ ることを示している。これらの酵素の而立熱性を測定してみると好熱菌部分の多い 2T2M6Tの方が4M6Tより熱に弱いことがわかった。したがって、N末からの60 -130残基がその特性に大きな寄与をしていると考えられた。両者のキメラ分子 は元の結晶と同形で結晶化でき、分子置換法、X-PLORによる精密化によって 構造を決めることができた。
キメラ酵素の構造解析から以下のような耐熱性に関する知見が得られた。 4M6Tと2T2M6Tの構造を10Tと比較するとフォールディングは同じであった。 構造の重ね合わせを行って、10Tとこれらキメラ酵素の間で顕著に異なる部位は、 構造の折り返しにあたるループ部分、50,81,110,147番の残基付近、 に見いだされた。このうち110番付近の構造変化が2T2M6Tに特徴的で、ここに. 生じている10Tからの変化、P110E/G111S、が構造的な不安定さの原因であると 考えられた。このことは、Tripleと名前を付けた2T2M6T-E110P/S111G/S113E で構造をもとの10Tのかたちに戻すことで耐熱性が回復したことから裏付けられ た。これら以外に、進化的耐熱化あるいは部位特異的変異で、キメラ酵素2T2M6T の耐熱性を改善するためにいくつかの酵素が作成された。S82RとI93Lがこれに該 当し、前者は分子表面のR82が水素結合で運動が制限されること、後者は空間にフ ィットするために取っていた不利な二面角の解除がおもな原因で分子が安定化し、 耐熱性が向上したと考えられた。
2 これからの構造解析
これまでに行った構造解析は、活性に関与するアミノ酸を決定するための構造 解析を除いて、耐熱性の解明を目的として静的に常温で行ってきた。研究の方法は、 天然型の酵素をキメラあるいは点変異で耐熱性の程度を下げること。劣化した耐熱 性の程度を、今度は回復することであった。この回復過程には、天然の10Tに戻せ ば良いという方向性に関する強い指標があった。この指標は、人工的に変異を起こ す方法と、進化的耐熱化と呼ばれる微生物を利用した人口馴化実験で、自然も同じ 方向性を選ぶことが認められた。これらの結果としてのIPMDHの構造解析は、耐 熱性の第一原理としての個々の構造変異の情報を与えた。耐熱性に関わるアミノ酸 早なにか?どのような機構が重要なのか?どこをのように変異させるのが容易か? しかし、次に来るべき第二段階として:なぜ耐熱性なのか?原子レベルのグローバ ルな機構、熱力学的な違い(エントロピーとエンタルピー)、蛋白質の変性機構の 違いの解明、安定化性のエネルギー的な記述を通して安定性と反応性の関係の明確 な議論が期待される。
詳細な議論に対して、構造生物学から与えることのできる示唆は、精密な構造 解析と蛋白質の存在の実体により近い構造解析に立脚している。Thermus IPMDH の構造解析においては、精密さにおいて、結晶の含水率が多く、分解能が2.4A程度 である。データの完全性が78%程度である。反射強度の測定精度が6%程度の誤差 をもつ。これらにより、温度因子の基底値が高い構造が与えられる。後者の存在実 体に近い構造解析では、Thermus IPMDHの活性が最も高い温度が60℃程度であ るが、結晶化および構造解析は、25℃程度程度の常温で行われている。これらの問 題が解決は、実験によるところが多いが、理論計算も大きな役割をはたすことと思 われる。
実験的な問題解決の方法として、Thermus IPMDHの精密な構造解析のため に、特に溶媒分子と蛋白質の関係を知るために、低温での構造解析を始めた。10T と2T2M6Tキメラ酵素を100Kと150Kで構造解析することにより、室温での構 造に比べて:格子定数が5%程度縮小すること。分子の熱膨張が10Tで0.2%、 2T2M6Tで0.3%減少すること。水の同定可能個数が10Tで61から296個へ、 2T2M6Tで79から243個へ増加すること。水のネットワークが10Tで12, 2T2M6Tで7、見つかった。温度因子の基底値は、10Tでガラス遷移点が認められ る低下を示し、2T2M6Tでは、顕著な減少はなかった。これらのことは、10Tの構 造よりもキメラの構造では依然として配位が揃わないことを示唆していると考えら れ、詳細な検討を進めている。後段の実体に近いとしては、結晶を急激に加温して、 ラウエ写真でデータを集める温度ジャンプ法を開発中である。放射光の利用により、 現在のところ、分解能2.3A、完全性43%、測定誤差9%程度の反射データで、分子 表面の親水性のアミノ酸が温度ジャンプで動くことが認められている。今後の放射 光実験で、さらに多くの情報が得られるように、努力したい。
謝辞
多くの方々にお世話になってここまで実験が進んだ.ここへ記して感謝致します。天然型のIPMDH の構造解析は今田氏(松下国際研)によるものである。重点研究芦田班には,温度ジャンプ装置の開 発を援助していただいた.温度ジャンプの開発は,リガクの協力があった.放射光実験においては, 筑波大の坂部氏,名古屋大の坂部氏,高エネ研PFの渡辺氏,鈴木氏やビームラインアシスタントの協力があった.
参考文献