1.はじめに
脊椎動物などの高等生物は外界からの有害物質に対して精巧な生体防御機構を備えている。細菌やウイルスなどの病原体が体内に侵入した場合、まずマクロファージなどの食細胞やNK細胞が非特異的な認識により病原体を攻撃して最初の防衛にあたる。この防衛線が突破されると、B細胞やT細胞などのリンパ球が特異的に病原体を認識して攻撃を行い、さらに活性化したB細胞は分化して抗体を産生してこれを体中に放出する。
最初にマクロファージが貧食作用を行うとき、T細胞も重要な働きをしている。T細胞はマクロファージなどにより提示された細菌抗原によって刺激され、総称してサイトカインと呼ばれる様々な種類の蛋白質因子を放出する。そしてサイトカインは他の免疫系細胞に対して情報伝達を行い、それによって生体防御機構の制御を行っている。そのなかにはマクロファージの走化活性を示すもの、マクロファージの活性化をおこなうものなどがある。1966年にDavidおよびBloom & Bennett らによって見い出されたマクロファージ遊走阻止因子(MlF)は最初に発見されたサイトカインである1,2)。マクロファージはMlFなどのサイトカインの働きによって感染部位に集められるのと同時に活性化される。活性化したマクロファージは、細菌を貧食して破壊する。
このように、結核菌などの細胞内奇生性細菌に対する生体防御機構においては、さまざまなサイトカインが相互に関係しながら制御を行っている。最近の報告では、MlFはT細胞の他にマクロファージや脳下垂体からも必要に応じて分泌され、グルココルチコイドと拮抗して免疫抑制や炎症作用を制御していると言われている3,4)。しかし、MIFはわれわれの体内で重要な役割を担っていると考えられて長い間研究されてきたにもかかわらず、その生理的な機能を理解するのに有効な報告はなされておらず、依然として生体防御のプロセスは明らかにされないままである。そこで我々はこの蛋白質の機能を、原子レベルの立体構造に視点をおいて研究することが必要であると考え、X線構造解析を行った。
2. 結晶の調整
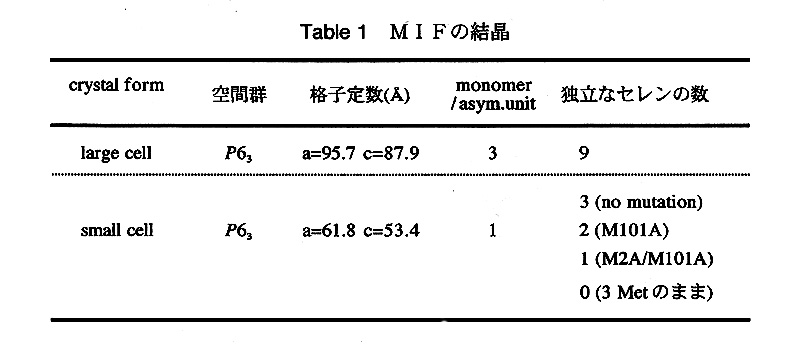
ラット由来のMlFは分子量12.3kDa、アミノ酸残基数114の蛋白質である。溶液中での実験からは2量体を形成していると考えられていたが結晶中では3量体であった。まず、大腸菌メチオニン要求株で蛋白質を発現させ、単量体中に3つあるメチオニン残基をすべてセレノメチオニンに置換した。セレノメチオニンを用いた解析法では、試行錯誤でのソーキングによって重原子置換体を作製する手間がなく、確実に位相決定可能な重原子置換体が得られることから非常に有力な手段として今では広く利用され始めている。しかし、ラットMlFのMAD法による構造解析は、初めのうちはうまくいかなかった。最初にできた結晶はVm値から推定して非対称単位中に1あるいは2個の3量体分子が存在し、独立なセレンの数は1×3×3=9個となる。その位置をパターソン関数から決定するのが困難であった。そのような理由から、解析の邪魔になりそうなメチオニン残基は遺伝子操作でアラニンに置換することにした。このとき、アミノ酸配列中の2、47、101番目のあわせて3つあるメチオニンのうち、どれを残してどれを変えたら良いのかが問題となったが、2次構造予測や疎水性残基の位置関係などから、なるべく全体の立体構造に影響がなく、分子表面にあって温度因子が比較的高そうなメチオニン残基を予想した。そしてそれらをアラニンに置換した複数の種類のミュータントを作製した。結果的にはそれらのミュータントでより格子の小さい結晶系が得られ、その条件でネイティブの蛋白質を含めていずれも同型な結晶が得られた(Table 1)5)。

3.回折実験
回折強度の測定は高エネルギー物理学研究所Photon FactoryのBL18Bで行った。セレン原子のK吸収端は0.98Å付近にある。蛍光X線を観測して吸収端の波長を確 認した。図1の矢印が示すように3波長でMADのデータを収集した。結晶はC軸方向に長細い形をしていたのでその方向に結晶を並進させることで、3つの異なる波長のデータを常温で1個の結晶で収集することが可能だった。このことで結晶による吸収の差などから起こる波長間での系統的な誤差がおさえられた。また、バイフィット対が1枚のIPフレームに測定されるようC軸を回転軸に一致させてワイゼンベルグ写真を撮った。セレンの数の異なる蛋白質の結晶が3種類あり(Table1)、全てに対して3波長で回折データ収集を行った。
4.構造解析
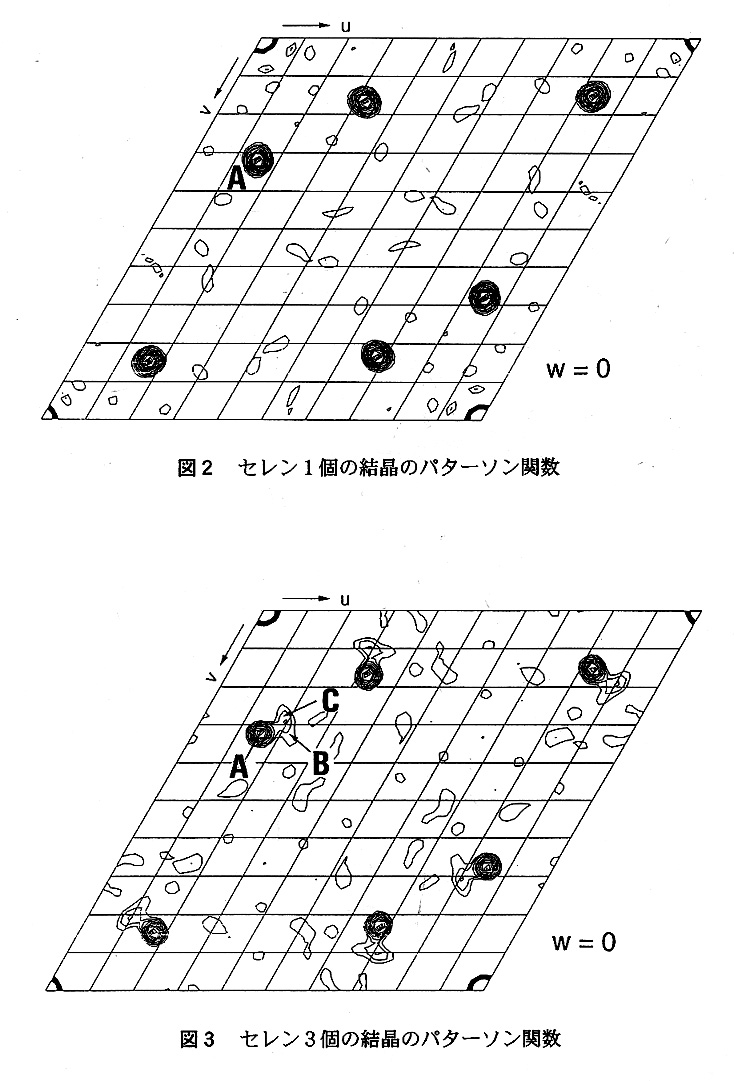
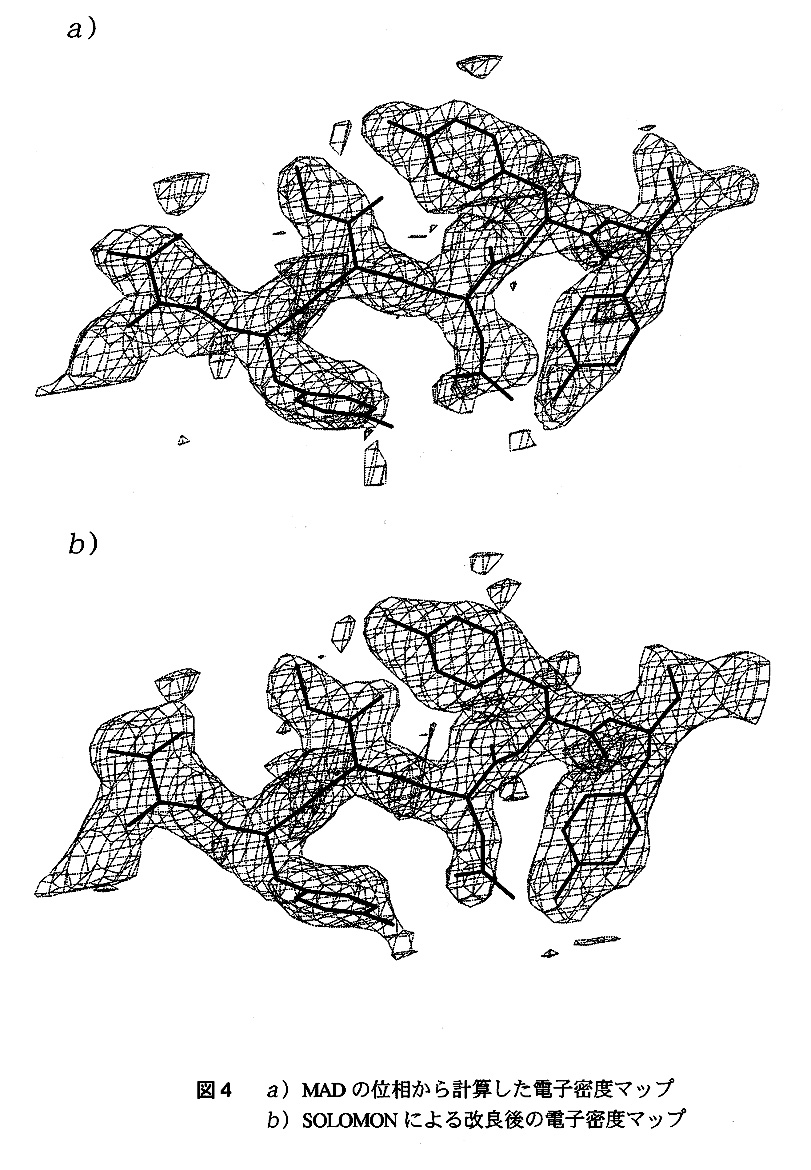
データ処理はDENZO6)とCCP47)を用いた。重原子位置の精密化と位相計算はMLPHAREを利用した。図2はモノマーに1個のセレン原子を含むミュータント結晶のパターソン関数を示す。この結晶は非対称単位中に一つのモノマーを含む。ハーカーセクションW=0面にはこのように20σ以上の明瞭なピークが現われた。それに対して図3にはミューテーションをかけないで3つのセレン原子を含む結晶のパターソン関数を示す。図2の結晶のセレンに相当するピークは明瞭であるが、それに比べて残りの2つのセレン原子のピークは不明瞭である。これはセレン原子の温度因子をそのまま反映している。図3のマップでも解釈は十分可能であり、そのデータからの位相情報の利用も可能ではあったが、この構造解析ではデータの質が一番良かったセレンを1個だけもつ結晶のデータのみを用いて行った。プログラムSOLOMONによる電子密度の改良後のマップは、最後までディスオーダーしていたC末端の11残基の領域を除いて非常に良質なものであり、プログラム○を用いて簡単にモデルの構築を行うことができた。(図4)
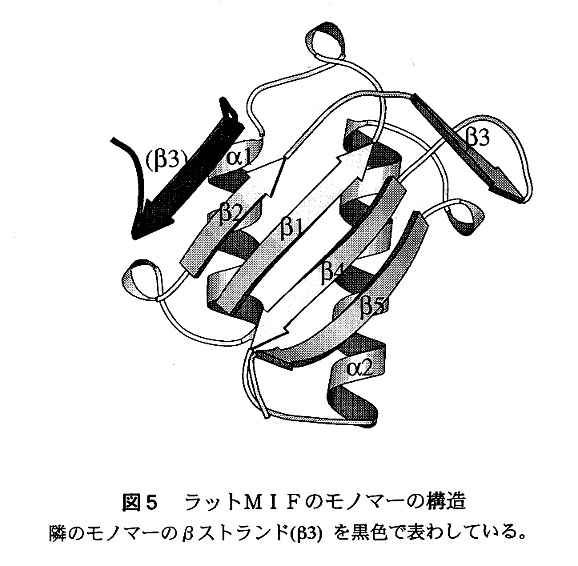
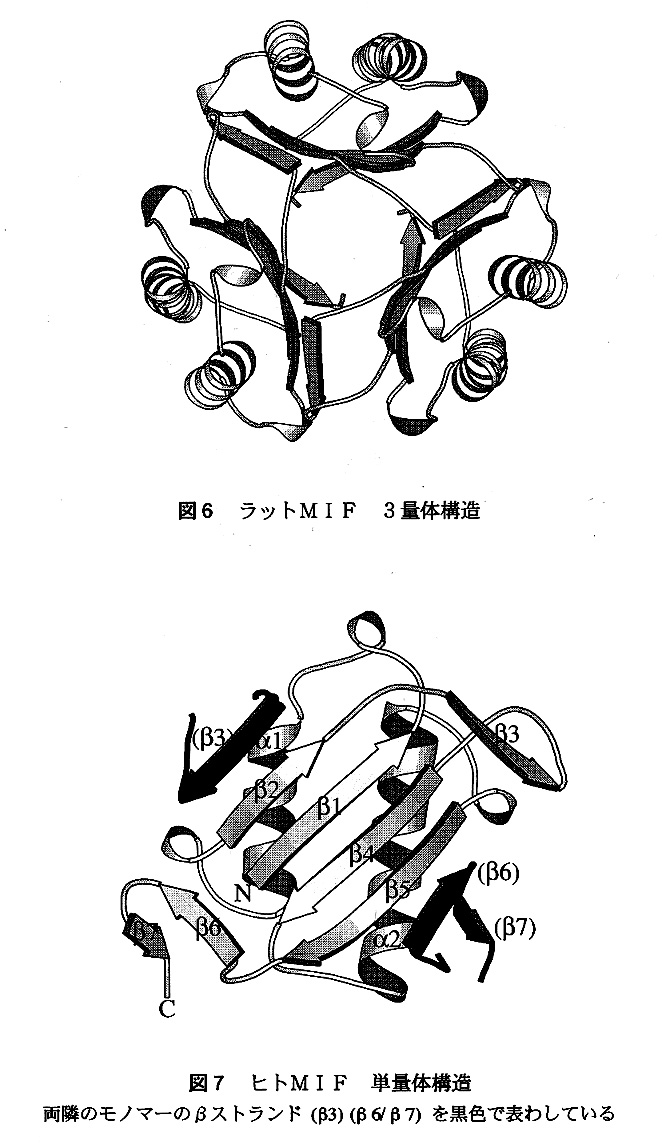
5.立体構造 図5にラットMlFのモノマーの構造をリボンモデルで示した。2つのβαβモチーフが疑似の2回軸で結ばれた形をしている。4本のβストランドは混合βシートを形成し、2本のαヘリックスは逆平行に配置している。さらにMlFの46〜50残基の領域には他の4本のβストランドに直交してもう1本のβストランドが存在する。このストランドは隣のモノマーのβストランドとの間で水素結合をつくり,ユニークな3量体構造を形成している(図6)。3量体の中心部分は5本のβストランドからなる3つのβシートによる“β−プリズム”があり、その周りに6本のαヘリックスが取り囲んでいる。この3量体は直径が約54Å,高さ約38Åの円筒状をしており、3回軸にそって水分子が通過できるほどのchannelが存在する。
さらにわれわれは、ラットMlFの座標をもちいて、ヒトMlFの立体構造を分子置換法で決定した9)。ヒトMlFの電子密度にはラットではディスオーダーしていて見えなかったC末端の11残基がはっきりと現われた。そしてそれらの残基はさらに3量体中のもう一方のモノマーとβシートを形成していた。すなわち3量体の中央にある3つのβシートは3つのモノマー全ての寄与により、合計7本のβストランドで形成されている(図7)。
6.異性化酵素としての機能
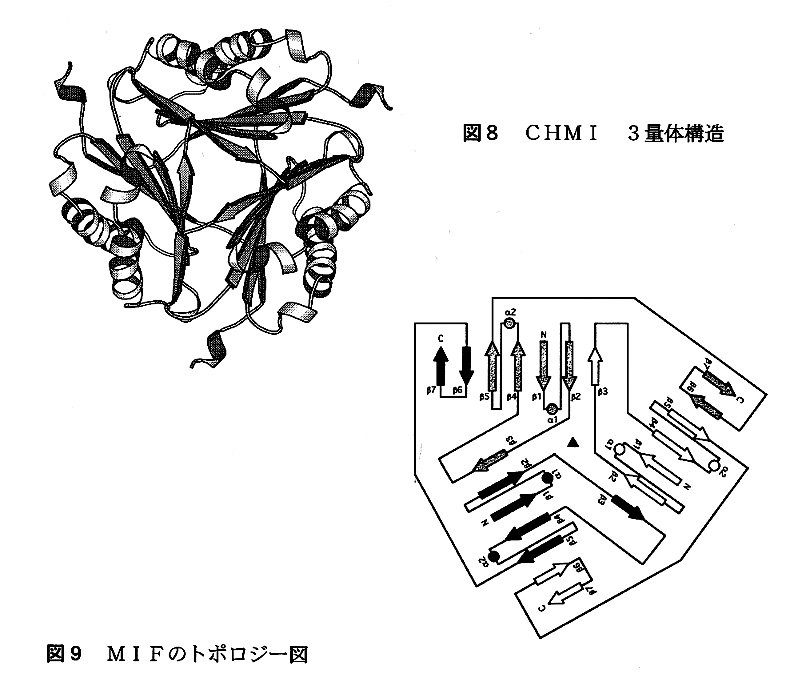
このような構造は一見インターロイキン−8や主要組織適合複合体HLA−A2といった蛋白質に類似しているように思われるが、これらは単量体として存在しフォールディングトポロジーも全く異なっている。SCOPやDEJAVUといったデータベースにもMlFのようなモチーフを見つけることができなかった。しかしながら、われわれはごく最近に構造解析された大腸菌のもつ不飽和ケトンの異性化酵素5-carboxymethyl-2-hydroxymuconate isomerase(CHMI)に同じモチーフを認めることができた(図8)10)。さらに、この酵素とMlFの1次構造を比較してみると、18%ほどの相同性が確認できた。MlFはこれまでに異性化酵素との関係は全く報告されていない。CHMIもMl Fと同様に3量体構造をとり、モノマ一間の相互作用の様式もほぼ同じである(図9)。モノマーに関して細かく見て見ると、CHMIは126残基であるためループの長さやαヘリックスの方向などに少しちがいがあるものの、フォールディングトポロジーは極めて似ている。
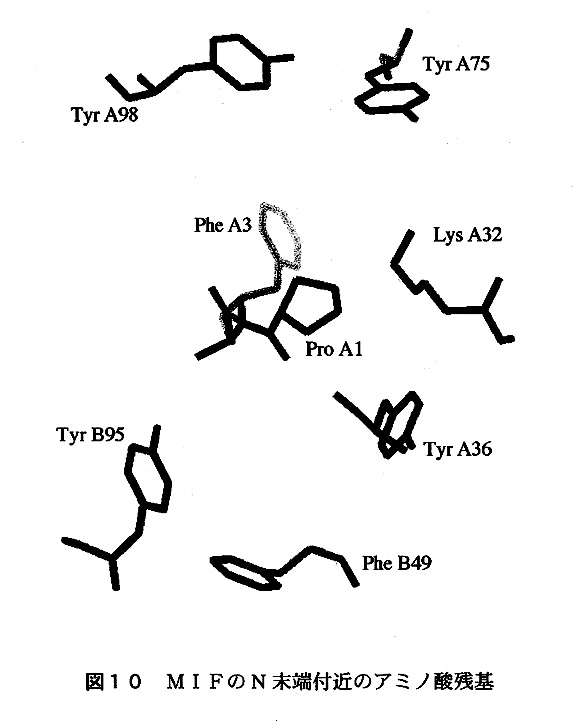
したがって、3次構造の類似性から、MlFの異性化酵素としての活性が推測される。その後の研究では予想どうり、ある基質に対しての異性化活性が確認できた。CHMIの活性部位はN末端付近の分子表面のポケットにあり、プロリンを触媒基としてアルギニンなどの負に帯電した側鎖が基質を安定化していると報告されている。N末端のプロリンはMlFでも保存されており、また、芳香環側鎖に囲まれたポケットが存在しているため、ここでCHMIとは異なる機構で反応を触媒している可能性が高い(図10)。MlFが異性化酵素と機能的あるいは進化的に関係があることはまちがいない。しかし、そのような活性が免疫抑制および炎症作用の調節というMlFのもつ主要な機能とどのような関わりがあるのかという点は今後の研究の課題である。
7.おわりに
この解析は鈴木守博士(高エネ研)、中川敦史博士(北海道大学大学院理学研究科)、西平順博士(北海道大学医学部)との共同研究である。また、放射光の利用にあたっては坂部知平博士と渡辺信久博士の協力を得て行われた。
文献