はじめに
最近の新聞およびテレビ報道でもおなじみのように、PCBによる環境汚染 が大きな社会問題となっている。PCBは、ポリ塩化ビフェニル(Poly Chlorhnated Biphenyl〉の略で、ビフェニルの水素のいくつかが塩素に置換したものである。この PCBは、水難溶性、不燃性、耐熱性、電気絶縁性などの優れた化学的性質のため、ト ランスの絶縁油など様々な工業的用途に用いられてきた。しかし、1968年のカネミ 油症事件をきっかけに、その毒性が明らかになった為に生産が中止され、使用も厳 しく制限されるようになった。現在ではPCBは使用されていないが、焼却して分解す ることが困難なため(不十分な温度で焼却すると、更に毒性の強いダイオキシンが 発生することが知られている)、過去に用いられたPCBは処理されることなく貯蔵さ れている。しかし、近年では安全な処理技術が開発されつつあり、貯蔵されている PCBを安全に処理することが近い将来可能になるであろう。
しかし、貯蔵されることなく環境中に放出されたPCBに関しては、なんら対策を講 じられずにいるのが現状である。現在までに環境中に放出されたPCBは75万トン以上 と言われ、その化学的安定性のため、分解されることなく環境中にとどまっている。 そして、魚介類や乳製品へのPCBによる汚染は引き続き進行しているのである。実際、 人体の脂肪組織には既にPCBの蓄積が認められている。これらは、PCBに汚染された 食物に由来するものである。更に、母乳には高濃度でPCBなどの有機塩素化合物が含 まれ、乳幼児への汚染が起こることも知られている。
このように、環境中に放出されてしまったPCBは、その生産や使用が中止された現 在でも、我々に大きな脅威を与えている。環境中に拡散しているPCBを一個所に集め て処理することは、事実上不可能であるため、如何にしてこれらのPCBを安全に分解 処理するかということは、今後の地球環境を考えるうえでも非常に重要な問題であ る。このような問いに対し、最近もっとも注目されているのが微生物の利用である。
PCBに関しては、1973年にAhmedらが初めてPCB分解菌を単離して以来、さまざま なPCB分解菌が単離されてきた1,2)。これらを用いれば、環境中に広く分布している PCBを安全に分解処理することも可能であろう。しかし、実際には、塩素置換数の多 いPCBは分解されにくいということや、分解系の酵素の誘導が十分でないなど、実用 化にはまだまだ多くの課題が残されている。これらの課題をクリアーするためには、 微生物の育種が必要である。我々の共同研究者である福田雅夫教授の研究室(筆者 と同じ長岡技術科学大学・生物系)では、長年にわたりビフェニル/PCB分解菌(以 下、単にPCB分解菌と記す)の単離やその分解系の酵素のクローニングを行ってきた。 そこで我々三井研究室では、福田研究室と共同でPCB分解菌のPCB分解代謝系を構成 する蛋白質群の立体構造解析を開始した。この共同研究では、立体構造に立脚した PCB分解代謝系酵素群の全容を解明することにより、立体構造に基づいたpCB分解系 の改良を行い、微生物による環境浄化法(Bioremediation)ヘの奇与をしようとして いる。更に、以下に記述するように、この酵素群の中には、立体構造や反応機構が 未知のもの(他の類縁の酵素を含めても)も多数含まれているので、これらの研究 を通じて酵素科学白体の進展にも奇与することを目指している。
PCB分解菌
我々は、共同研究者である福田教授らのグループによって単離された
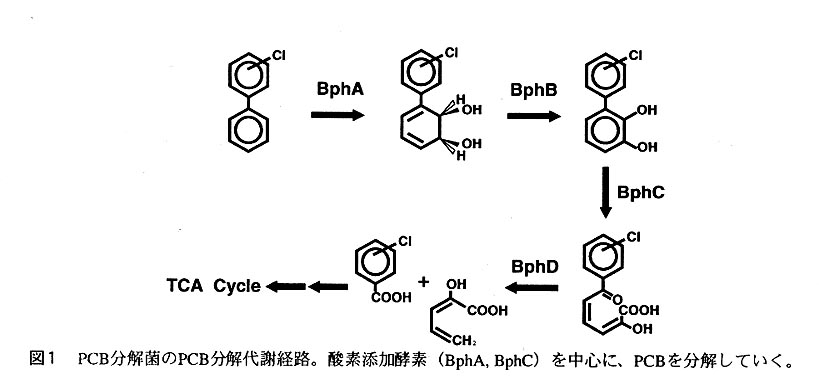
PCB分解菌であるPseudomonas sp. strain KKS102およびRhodococcus sp. strain RHA1の酵素群について研究を行っている。これらの菌は、図1に示すようなPCB分解系を持っている2。

まず、PCBを含むビフェニル(biphenyl)の誘導体は、BphAという初発酸化酵素に よって水酸基を付与される。次にBphBにより脱水素反応をうけ、BphCによって芳香 環の開裂をうける。その後、BphDにより安息香酸と2-hydroxypenta-2,4-dienoateに分解され、更にBphE,F,G,という酵素群によってアセチルCoAにまで分解され、TCA cycleへと入っていくのである。現在我々は、これらの酵素について、X線結晶解析 の手法を用いて立体構造解析を行っている。既に、BphC、BphDに関しては立体構造 を得ている。本稿では、もっとも解析の進んでいるBphCを中心に最近までに得られ た知見を述べる。
BphC酵素について
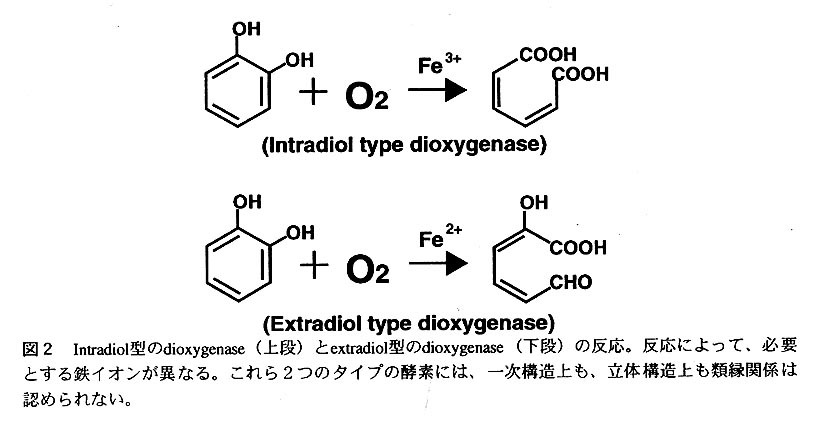
Dioxygenase(2原子酸素添加酵素)は、2つの酸素原子を基質 に導入する酵素で、戦後まもなく全く新しい酵素として早石修博士により世界に先 駆けて発見された。当時は、空気中の酸素分子を取り込んで酸素原子を直接基質に 付加するという生体酸化反応は知られておらず、新しい生体反応を触媒する酵素と して注目されてきた。我々が研究を行っているBphC酵素は、カテコール環を開裂す るdioxygenaseで、dioxygenaseの中でも最も歴史の古い酵素である。現在までに、この 類いのdioxygenaseには大きくわけて2つの種類が知られている。一つは、カテコール 環の2つの水酸基の間を開裂するintradiol型のdioxygenaseで、活性中心に3価の鉄イ オンを必要とする酵素である(図2上段)。もう一方は、カテコール環の2つの水 酸基の外側を開裂する酵素で、活性中心に2価の鉄イオンを必要とするextradiol型の dioxygenaseである(図2下段)。両酵素とも活性中心に鉄イオンを含むが、上記のよ うに2価と3価という違いがある。そしてこれに対応して、反応機構もin比adio1型と extradiol型で根本的に異なっていると考えられている。つまり、in血diol型の酵素では、3価の鉄イオンを利用して、基質であるカテコール(およびその誘導体)を活性化 し、反応を進行させるのに対し、extradiol型では、酸素分子を活性化し、その結果反 応が進行すると考えられている(これらはいずれも分光学的研究から得られた結果 である)。
立体構造解析に関しては、intradiol型のdioxygenaseは1988年にLipscombらのグルー プにより立体構造が決定されている13〉。一方、extradiol型酵素の結晶化は古くから行 われていたにもかかわらず、良質の結晶を得ることが困難で、その立体構造も長い 間不明のままであった。しかし、幸運にも我々の用いたPCB分解菌(Pseudomonas sp. strain KKS102)から単離したBphC酵素は、非常に良質な結晶になることが判明し (放射光を用いた場合は1.2Å程度の分解能が得られている〉、X線結晶構造解析を 行うことができた。
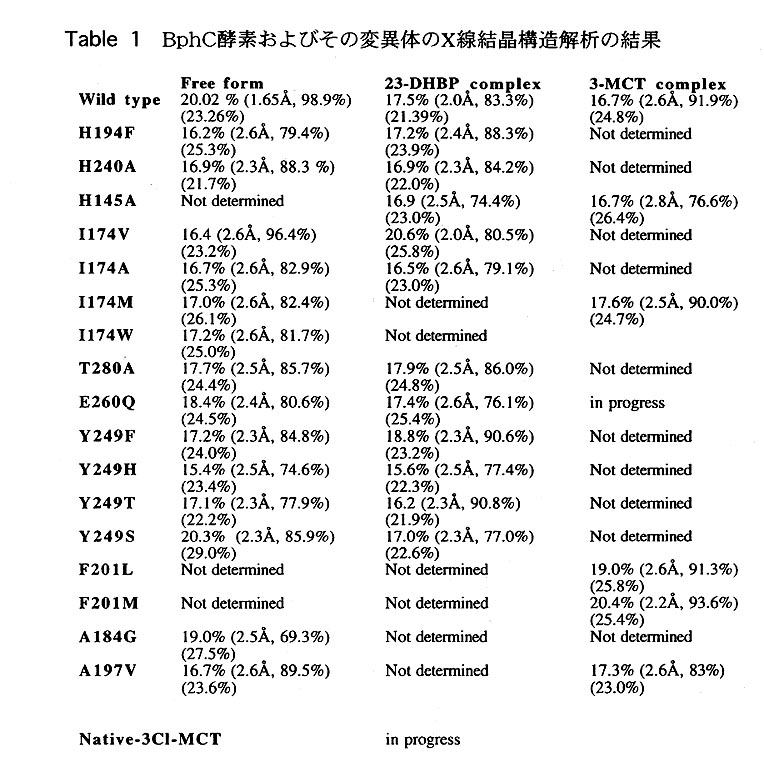
本稿で取り上げるKKS102由来のBphC酵素3)は、分子量32KDaのサブユニツトが8つ集まってできる総分子量が250KDaの巨大な酵素である。各サブユニットに一つの活 性部位を持ち、活性部位には他のextradiol型のdioxygenaseと同様に2価のノンヘム鉄を 有している。我々は、1995年に世界に先駆けてこの酵素の立体構造を重原子同型置 換法により決定した4.5.6)(同じ年にPurdue大のBolinらのグループも我々のBphC酵素と 約60%のホモロジーを持つ酵素の解析に成功している14))。これに加え、基質である2,3-DHBP(2,3-dihydroxybiphenyl)及び3-MCT(3-methylcatechol)との複合体の立体構造の決定も行った6)。また、この酵素の基質特異性、反応機構の解明を行うため、福田研究室では現在までに約50種類の変異体を作成し、速度論的データの測定を行ってい る。その内の17種類については、三井研究室においてX線結晶構造解析により立体 構造の決定を行い、活性と構造の両面から解析を進めている。現在までに行った、 結晶学的解析のまとめをTable 1に示す。
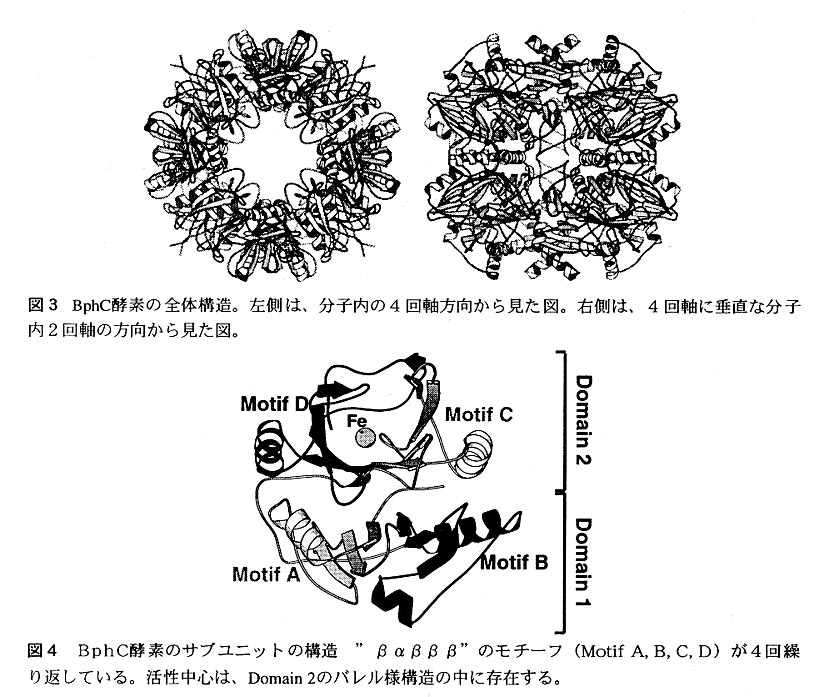
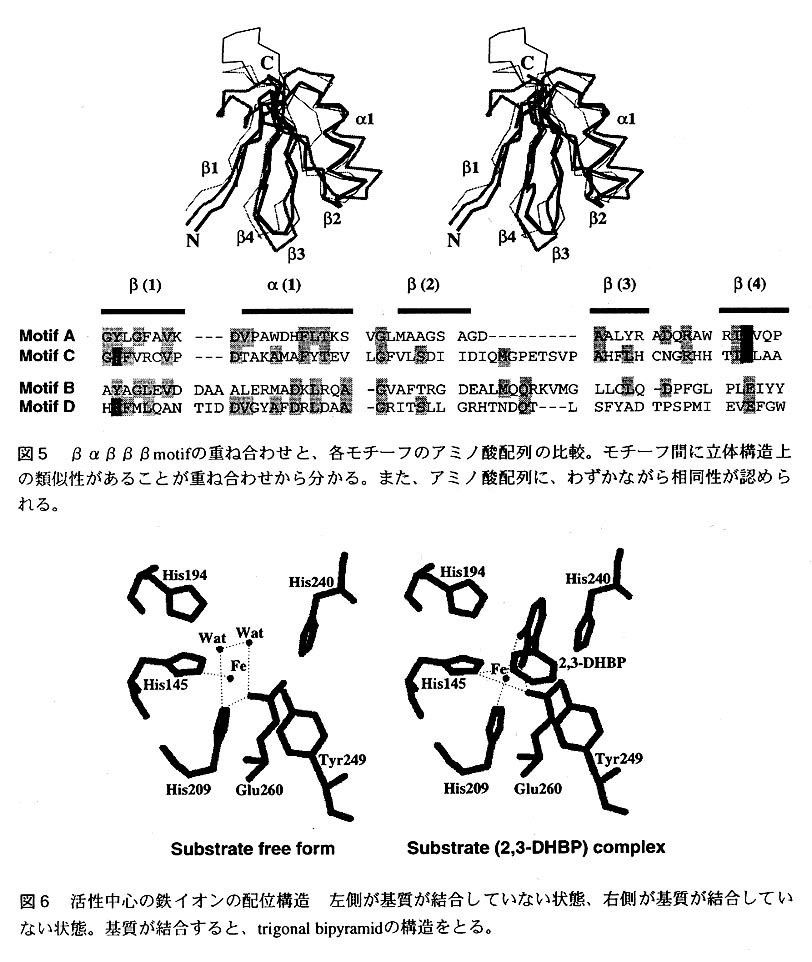
BphC酵素の立体構造5,6,7)図3に示したのはBphC酵素の全体構造である。ドーナツ状の4量体が2段に重なるようにして8両体を形成しており、分子全体としては点 群422の対称性がある。分子の直径は約100Åで、中心には最大部分で25Åある穴が開 いている。BphC酵素のサブユニットの立体構造を図4に示す。BphC酵素のサブユニツ トは2つのドメイン(N末側のDomain 1とC末側のDomain 2)に分かれており、活性 部位はC末側のDomain 2のバレル様構造の内側に存在する。各ドメインは、"βαβ ββ"型のモチーフ(図中のMotifA,B,C,D)が2回繰り返された構造を持っている。 このため、各ドメインはよく似た立体構造となっている。これらのモチーフの立体 構造を最小自乗法により比較してみると、2次構造部分のCα原子のみを用いて計算 した場合、各モチーフ間のrmsdは、すべて2Å以下となり、互いによく似ていること が明らかになった(図5の上段参照)。また、各モチーフ問のアミノ酸配列を比較 してみると、非常に弱いながら(20%前後)アミノ酸配列の相同性が認められた(図 5の下段参照)。これらのモチーフ間に見られる立体構造およびアミノ酸配列上の 類似性は、この酵素が2度にわたる遺伝子の重複により進化してきた事を強く示竣 している。
活性中心の鉄イオンは、His145,His209,Glu260および二つ(もしくは一つ)水分 子と配位しており、square pyramidの配位構造をとっている(図6(左))。これら の残基は類縁酵素間で完全に保存されており、類縁酵素では共通の活性部位構造を 持つことが明らかである。基質複合体では、基質の結合に伴って水分子が鉄から外 れ、基質の2つの水酸基が鉄に配位することが基質複合体の結晶構造より示された。 このときの配位構造は、trigonal bipyramid(図6(右))になっていた。
基質の結合8)
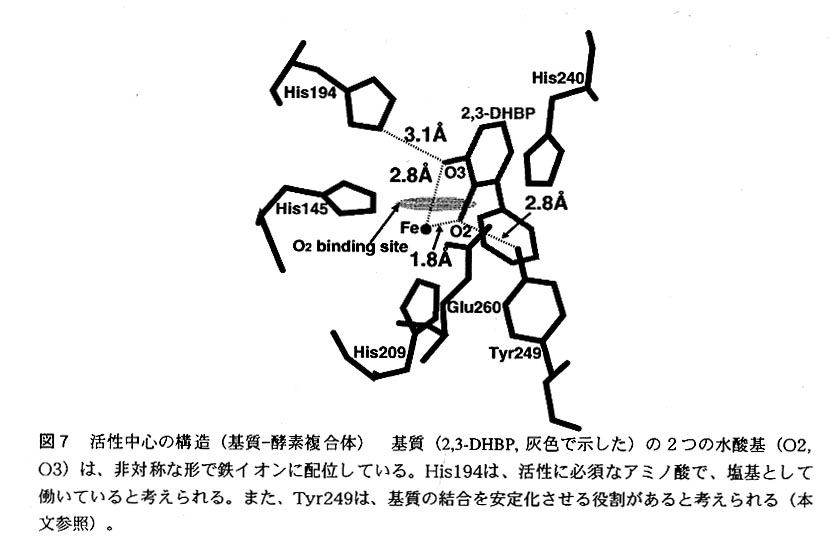
基質複合体の立体構造解析より、基質の2つの水酸基の鉄への配位は、 図7に示したような非対称な形であることが明らかになった。カテコール環の2位 の水酸基の酸素原子と鉄との距離は1.8Åと非常に短く、この水酸基はイオン化して 鉄イオンに配位していることが示唆された。これに対し3位の水酸基は、鉄との距 離が2.8Åとなっており、イオン化せずに鉄イオンと配位していると考えられた。こ の様な非対称な形をした基質の配位形式は、Lipscombらにより提唱されている反応機 構(図8参照)と一致する。一方、基質結合ポケットの形状を詳細に調べたところ、 基質と基質結合ポケットの形状には非常に高い相補性があり、上に述べたような非 対称な配位様式以外はとりにくいことがわかる。つまり、基質ポケットの形に沿う ように基質が結合すれば、おのずから上記のような非対称な配位構造をとるように 基質ポケットは設計されているといえる。
疎水相互作用が基質の結合に重要である8)種々の変異体の立体構造解析を進めてい くうちに、基質の基質結合ポケットヘの結合には、基質の水酸基の鉄への配位結合 が必須ではないことがわかってきた。活性中心の鉄イオンに配位しているHis145を 州aに変えた変異体では、活性中心の鉄イオンは無くなっている。しかしこの結晶を 基質である2,3-DHBP溶液にソーキングし構造解析を行ったところ、基質に対応する 明確な電子密度が観察された、これとは対照的に、基質とスタッキング相互作用を しており、基質結合ポケットの形状を作る上で重要と考えられるHis240を州aに変え た変異体では、ソーキングによって結晶中の変異BphC酵素に基質を導入することは できなかった。また速度論的解析からも、この変異体(His240Ala)ではKm値が大幅 に上昇するという結果が得られている。これらの結果より、基質の結合に重要なの は、基質と酵素間の疎水相互作用であることが示唆された。
以上のことを総合的に考えると、基質は疎水相互作用によって基質結合ポケット に結合し、基質と高い相補性を持つ基質結合ポケットによりポケット中での配向が 決められる。そして、鉄イオンとの配位に際しては、2位の水酸基(O2)が特異的に イオン化すると考えられる。
Tyr249は、基質の結合を安定化する
Tyr249も、類縁酵素問で完全に保存されてい る残基で(Table 2参照)、活性中心の鉄イオンと鉄に配位した水酸基の近傍に存在 することから、活性に関与すると考えられた。そこで、種々の変異体を作成し、活 性の測定、立体構造の決定を行った。その結果、Tyr249を他のアミノ酸に変換した変 異体では、いずれも活性が保持されているものの、どの変異体においてもKm値が上 昇し、基質との結合が弱くなっていることが示唆された。更に、これらの変異体に ついても基質との複合体の結晶構造を解析した結果(Table1参照)、いずれの場合も wild typeの基質複合体に比べて、基質の温度因子が有意に上昇していることが観察さ れた。これらの結果を考え合わせると、Tyr249は酵素の触媒機構に必須ではなく、基 質の結合を助ける働きを持っていると考えられた。

酸素の結合位置6,7,8)
図8に示すように、BphC酵素の反応では活性化された酸素分子 がC1の位置を攻撃することにより、部位特異的な芳香環の開裂がおこる。基質複合 体の立体構造に基づいて詳細な検討を行うことにより、酸素分子の結合部位(oxygen binding cavity)を一カ所に限定することができた。この酸素結合部位は、基質のカテ コール環に対し、His240の反対側に存在すると考えられ(図7参照)、溝状の形をし た酸素結合部位はカテコール環のC1-C2結合にほぼ平行になっていることがわかった。 この溝状の酸素結合部位の立体構造的な制約により、活性化された酸素がC1を攻撃 するという反応の位置特異性が生じると考えられる。更に、酸素結合部位を上の様 に仮定すれば、C1の位置が正四面体構造をとる中間体(図8(b)参照)が、基質結合 ポケット内でどのように存在するかについてもうまく説明することができる。
His194は酵素反応に必須である8)
基質結合ポケットを形成するアミノ酸残基のう ち、基質のカテコール環に近位置にあるものは、類縁酵素問で完全に保存されてい る残基が多い。これらの中に、触媒活性に必須なアミノ酸残基があると考え、種々 の変異体を作成し、酵素活性の測定を行った。その結果、類縁酵素間で完全に保存 されている残基であるHis194(Table 2参照)を他のアミノ酸に変異させた変異体酵素 は、完全にその活性を消失することが明らかになった。更に、His194Phe変異体の結 晶構造を決定したところ、活性部位に構造上の変化は見られなかった。これより、 His194は酵素活性に必須であることが明らかになった。Lipscombら9)が分光学的実験 をもとに提唱している反応機構によれば、このヒスチジンは3位の水酸基の水素を引 き抜く塩基に相当すると考えることができる(図8参照)。また、この水素を引き 抜く過程が、BphC酵素の行う酸素添加の反応に必須の過程であることが示された。
BphD酵素について
BpHD酵素は、BphC酵素によって開裂された芳香環化合物を更に加水分解する酵素である。この酵素は、1ipase,ha1oalkane dehalogenase等のhydrolase と類縁関係にあると考えられている。実際に、我々によって解明されたBpllD酵素の 立体構造も上記の酵素と高い相同性があり、上記の類縁関係が確認された。 一方、PCB分解という観点からみると、BphD酵素の触媒する加水分解反応がPCB 分解代謝の律速段階になっているケースがあることが知られている。BphD酵素の基 質特異性が狭いために、BphC酵素によって芳香環の開裂反応を受けた中間代謝産物 が菌体内に蓄積し、PCB分解が思うように進まなくなってしまう。そのため、BphD 酵素の基質特異性の改善や反応性の向上は、高いPCB分解能を有する分解菌の育種に は欠かせない。
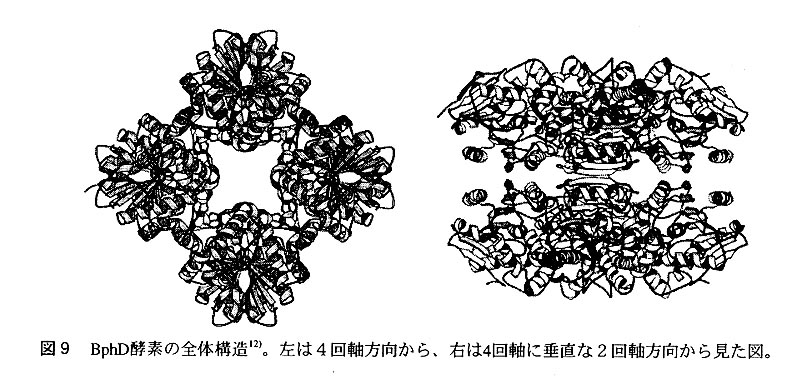
今回我々が重原子同型置換法によって構造を決定したBplmD酵素は、共同研究者で ある福田教授のグループにより単離された強力なPCB分解菌Rhodococcus sp. strain RHA1由来の酵素である10)。サプユニット当たりの分子量は32KDaで、ホモオクタマー (8量体)を形成しており、全体としての分子量は約250KDaである(図9を参照)。
BPhD酵素の立体構造11,12)
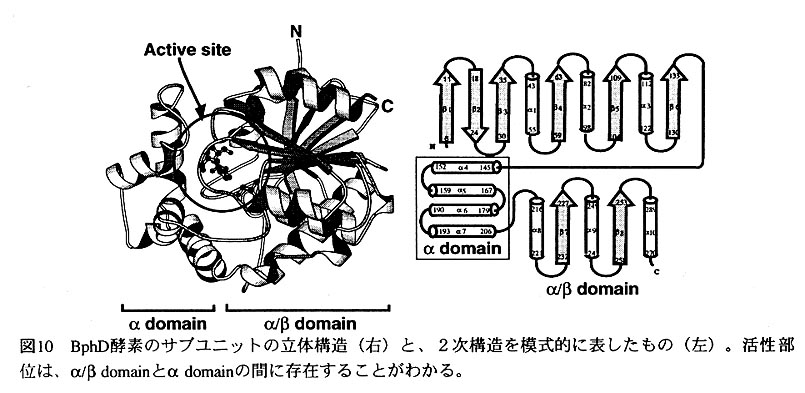
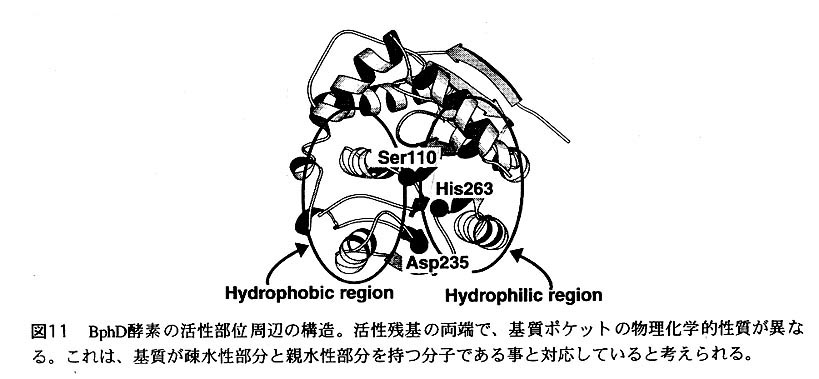
図9にBphD酵素の立体構造を示す。サブユニットは2つ のドメイン(α/β domainとα domain)からなっている。この構造は、現在までに立体 構造の決定されているIipaseやhaloalkane dehalogenaseと、非常に類似性の高いものであっ た(アミノ酸配列上はほとんど、ホモロジーは見られなかった)。活性部位は、2 つのドメインの中間に位置しており(図10参照)、完全に分子内部に埋もれていた。 活性残基は、Ser110、Asp235、His263で、他のhydrolaseやserdneproteaseと同様に、い わゆるcatalytic triadを形成している。
活性部位の構造
既に述べたようにBphD酵素の活性部位は、α/β domainとα domainの 間に存在する。活性部位は、完全に分子内に埋もれており、Ser110,Asp235,His263 の3つの触媒残基も完全に分子内部に存在している。これらの残基は、大きな基質 結合ポケット内のほぼ中央に存在している。基質ボケットを形成するアミノ酸残基 の性質は、これらの触媒残基を境にはっきりとした違いが見て取れる。すなわち、 3つの触媒残基からみて基質の入り口側は(図11では、左側の部分)、疎水的アミ ノ酸が基質ポケットを形成している。これに対し、触媒残基より奥の基質ポケット は(図11では、右側の部分)、親水的アミノ酸によって形成されていることが明ら かになった。これは、疎水的部分と親水的部分を持つ基質の性質と対応していると 考えられる。つまり、基質のベンゼン環部分は基質ポケットの入り口側に結合し、 基質のカルボン酸および水酸基部分は基質ポケットの奥に結合すると考えられる。 現在、さらに詳しいBphDの反応機構を解析するために、阻害剤を用いた実験を進 めている。
BPhA酵素について
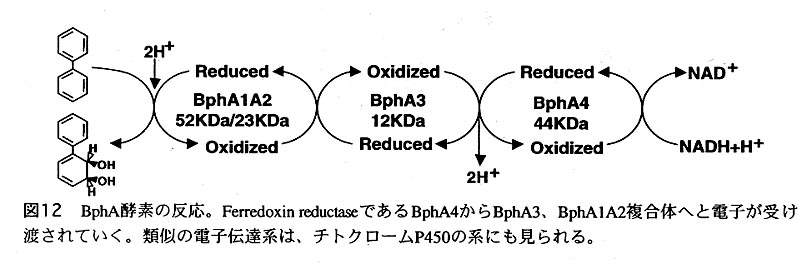
BphA酵素は、ビフェニル/PCB分解系における初発酸化酵素で (図1)、ベンゼン環に2つの水酸基を導入するdioxygenaseの一種である。この酵素 は、チトクロームP450等と同様に、電子伝達系をもつマルチコンポーネントの酸化 酵素で、BphA1,A2,A3,A4の4つのコンポーネントよりなっている。我々の問題に しているBphA酵素は、図12に示すような電子伝達系を持つ事が知られている。 BphA4は、NADHを必要とするおferredoxin reductaseで、アミノ酸残基数411からなる分 子量44KDaの蛋白質である。このBphA4から電子を受け取るのがBphA3のfe灯edoxinで ある。このferredoxinはterminal oxidaseであるBphA1A2複合体に電子を供給する。 BphA1A2複合体はdioxygenaseの本体で、分子状酸素を取り込んで、基質であるPCBや biphenylに水酸基を付加する。活性中心を含むBphA1には、いわゆるRieske typeの 2Fe2Sの鉄硫黄クラスターとノンヘム型の鉄イオンが1つずつ存在しており、これら と酵素活性がどのような関連を持っているかが注目されている。また、現在のとこ ろ、BphA2の機能は不明である。現在、我々はこれらの酵素の立体構造を解明するた めの実験を行っている。
BphA1A2複合体の精製・結晶化
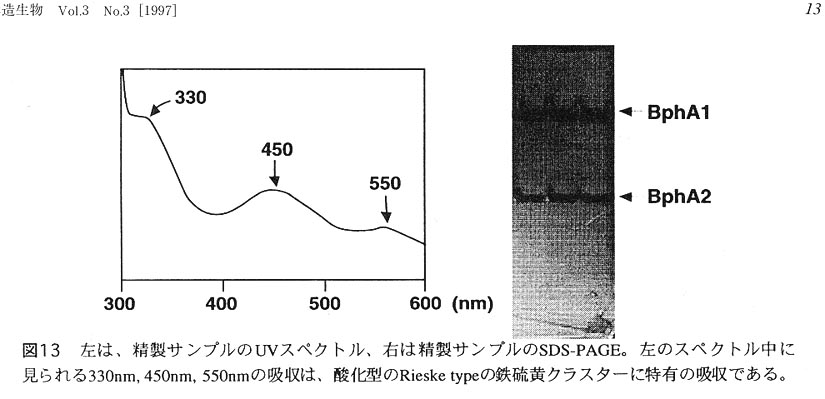
Pseudomonas sp. strain KKS102由来のteminal oxygenaseであるこの酵素はBphA1(Large subunit, αsubunit;458残基)とBphA2 (Small subunit,βsubunit;193残基)が3つずつ会合してできたα3β3の構造をとっており、総 分子量は225KDaである。各々の large subunit(BphA1)は、2Fe2Sの鉄硫黄クラスター と、ノンヘム型の鉄を一つずつ含んでいる。現在までに、類縁酵素を含め、この類 いのdioxygeaseについては立体構造はおろか結晶化も報告されていない。そこで本酵 素の結晶解析を行うために、Pseudomonas sp. strain KKS102由来のBphA1A2複合体を 大腸菌により大量発現し、精製を行った。イオン交換カラムとハイドロキシアパタ イトカラムによりこの酵素をSDS-PAGEにおいてほぼ単一バンドにまで精製できた (図13)。このサンプルを用いて結晶化を行ったところ、非常に小粒ながら(0.1x 0.1x0.1mm3)結晶を得ることができた。現在この結晶の質を改善するために、結晶化条件の最適化を行っている。
BphA4の立体構造解析
BphA4は、初発酸化酵素の電子伝達系における最初のコン ポーネントである。FADを含むフラビン酵素で、更にNADHを必要とする。分子量は、 44KDaである。現在この酵素の大腸菌による大量発現、精製に成功し、良質の結晶も 得ている。この結晶は、実験室内のX線回折装置を用いた際には、2.5Å分解能程度 までの反射を与える。現在、重原子同型置換法による構造決定を行うため、重原子 同型置換体の探索を行っている。
おわりに
本研究は、1993年に故矢野圭司教授のグループ(現在は、福田雅夫教授) との共同研究としてスタートした。それ以来、多くの先生方や学生の皆さんの協力 でようやくPCB分解系の酵素群の構造決定とそれに基づいた酵素の機能解析が軌道に 乗ってきた。今後は、初発酸化酵素BphAのすべてのコンポーネントの構造決定を始 めとする酵素系の全容解明、更にはこれら酵素の誘導の機構等も立体構造を元に論 じていきたいと考えている。
結晶構造解析は、筆者の属する長岡技術科学大学・生物系の三井幸雄教授のグルー プで行われた。実際に構造解析に携わった学生は(卒業生を含む)、表記のN. Na祖1dhagopa1、杉本敬祐、のほか、杉山和之氏、成田裕樹氏、郡司哲也氏、山田高広 氏、岡野みつ代氏、櫻井宣之氏、神田健司氏、外園成和氏である。BphDの解析は、 主に著者の一人Nandhagopalによって行われた。また、pCB分解菌の単離、遺伝子の クローニング、発現系の構築、および変異酵素の作成とその活性の測定は、長岡技 術科学大学・生物系の福田雅夫教授のグループ(福田雅夫教授、金原和秀博士(現 JR端総研)、政井英司博士、山本健氏、田口和之氏、西崎智子氏)によって行われた。 本稿で取り上げたBphCの変異体酵素の作成と活性測定の大半は西崎智子さんの仕事 である。
最後に、X線構造解析に際して数々の討論を頂きました野中孝昌助教授、変異蛋白 質の作成等、蛋白質工学的実験について数々の討論を頂きました福田雅夫教授、政 井英司博士に感謝します。また、高エネルギー物理学研究所でのデータ測定の際に お世話になりました、坂部知平名誉教授、渡辺信久博士、鈴木守博士に感謝します。
【引用文献】