1.はじめに
DNAは,熱,紫外線,ストレス等で容易に変異を受ける.この様な変異(化 学変化)をうけたDNAは生態系の遺伝情報維持に支障を来す恐れがあるが,こうし た損傷DNAを修復する酵素群が生体内には存在する.これらはDNA修復酵素と呼ば れている.光回復酵素(photolyase)はDNA修復酵素の一つで,紫外線によってDNA に生じる損傷の一つであるピリミジン二量体(チミンの二量化,図1)に結合し, その後に吸収する光のエネルギーを用いて元の単量体に修復する.このDNA修復は 光回復と呼ばれる.
ラン色細菌由来の光回復酵素は,分子量約5万のアポタンパク質とフラビン 骨格をもつ2種の補欠分子からなっている(1). 光回復酵素はDNAに生じる損傷DNA のピリミジン二量体に結合した後,吸収した光のエネルギーをトリガとして二量体 から単量体への修復を行う.興味深いことに修復時に用いる波長が,長波長型の光 回復酵素(〜440nmを吸収)と短波長型のもの(〜380nm)に分類することができる. 本研究で対象としたラン色細菌由来のものは長波長型(2),一方,大腸菌由来のもの は短波長型に属する(3). これは,2種の補欠分子フラビンの構造の違いに起因して いる.どちらの酵素も還元されたFADを第1の補欠分子として共通にもっているが, 第2の補欠分子の種類が異なることにより,長,短波長型の違いが生じている.す なわち,長波長型ではデアザフラビン型補欠分子を,短波長型ではプテリン型補欠 分子を有している.この第2の補欠分子は光を吸収して,そのエネルギーを還元型 FADに渡す集光補欠分子であり,このエネルギーを受けて触媒補欠分子であるFAD によるピリミジン二量体の修復が行われる.
最近,プテリン型の集光補欠分子を持つ大腸菌由来の酵素の結晶構造が Deisenhoferのグループによって解明されたが(4),一方,我々はデアザフラビン型補 欠分子を持つラン色細菌由来の酵素の結晶構造解析に成功した(5). その結果,驚く べきことに,全体のフォールディングには高い共通性があるにもかかわらず,集光 補欠分子の結合部位が全く異なっていることを見い出した.これは集光補欠分子か ら触媒補欠分子(FAD)ヘのエネルギー移動が両酵素で異なっている点から極めて興 味深い.

2.ラン色細菌由来光回復酵素のX線構造解析
ラン色細菌由来の光回復酵素の結晶は,硫酸アンモニウムを沈殿剤として蒸 気拡散法により得られた.中性付近のpHで得られた結晶は,柱状結晶が集まったク ラスター状態であったが(6),EDTAを添加することにより,六角柱状の単独の結晶と なった(7). いずれの結晶もPFのBL6Aピームラインにおいて巨大分子用ワイゼンベ ルクカメラ(坂部カメラ)を用いてX線回折実験を行うことができたが,クラスタ 一状柱状結晶(クラスターから切り出して用いた)が1.8Å分解能を与えたのに対し て,六角柱状の結晶は3.0Å分解能程度の回折点しか観測することができなかった. したがって,柱状結晶を用いて構造解析を行った.この柱状結晶は,正方晶系に属 し,空間群P43212,a=b=91.17Å,c=135.54Åで,非対称単位中に一分子の光回復酵 素を含んでいた.回折強度データの測定もPFで坂部カメラを用いて行った.3種の 重原子を用いた童原子同型置換法により,1.8Å分解能の結晶構造解析を行うことが できた.
3.ラン色細菌由来光回復酵素の立体構造
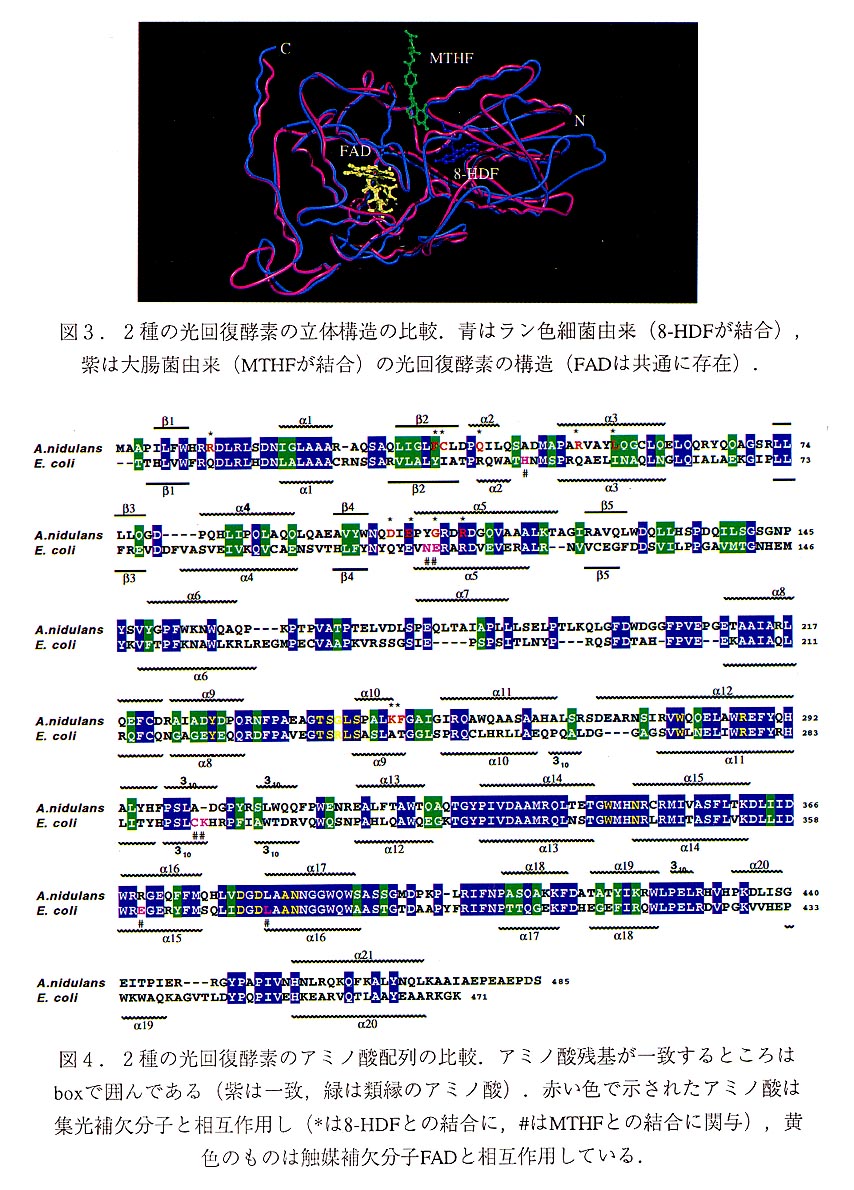
ラン色細菌由来の光回復酵素の全体構造を図2に示す.分子は二つのドメイ ンに分かれる.N末端側はいわゆるRossmannfoldをとっているα/βドメインであり, 一方,C末端側に多くのヘリックスからなるヘリカルドメインが位置している.α11 ドメインには,集光補欠分子である8-ヒドロキシ-5-デアザフラビン(8-HDF)が, 一方,ヘリカルドメインには触媒補欠分子であるFADが結合していた.分子全体の フォールディングは先に決定された大腸菌由来の光回復酵素と非常に良く似ており (図3),両者のCα炭素のr.m.s deviationは2.2Åであった.立体構造上の違いはラン色細菌由来の光回復酵素にのみ,N端側のα/β構造に付随してもう一本α-ヘリックスが存在していることであった.
図4にはラン色細菌由来,大腸菌由来の光回復酵素のアミノ酸配列を示して ある.立体構造上の相同性と相まって2つの酵素の一次構造は良く似ており,39% が同一残基,13%が性質類似の残基であった.
活性発現に必要な補欠分子であるFADは,いずれの光回復酵素でもほとんど 同一の位置に存在し(図3),2つの光回復酵素のFAD分子でのr.m.s. deviationは 0.24Åであった.また,FAD分子と相互作用してこれを保持するアミノ酸残基も両者 の酵素で非常によく一致していた.しかしながら,α/βドメインの集光補欠分子は, 以下に示すように,両者で全く異なった位置に結合していた.
4.集光補欠分子の認識
光回復酵素の集光補欠分子は吸収した光エネルギーを触媒補欠分子である FADに伝達する.触媒補欠分子によるピリミジン二量体の修復反応にはこのエネル ギーが必要である.先に示したように,ラン色細菌由来の光回復酵素は集光補欠分 子として8-HDFを,大腸菌由来の光回復酵素はプテリン型補欠分子である5,10-メテ ニルテトラハイドロ葉酸(MTHF)を有している.ラン色細菌由来の光回復酵素の結晶 構造が明らかとなったことで,2種の光回復酵素における集光補欠分子,8-HDF(ラ ン色細菌)ならびにMrHF(大腸菌)の結合位置は,全く異なっていることがわか った.いずれもN末のα11ドメインに結合しているが,8-HDFはMrHFに比べて12Åも 分子の内側に入り込んだ位置にある.このα/βドメインのフォールディングは2つの 酵素でほとんど一致しており(図5),この部分のアミノ酸配列についてもβ1から α6まで非常に相同性が高い(図4).すなわち,三次元構造がほとんど同じで,し かもアミノ酸残基の相同性が極めて高いにもかかわらず,両者それぞれで集光補欠 分子と相互作用するアミノ酸を少しずつ変えることにより,異なる集光補欠分子を 認識していることが示されたのである.上に述べた集光補欠分子と相互作用するア ミノ酸残基を具体的に図6で示すと,たとえば大腸菌のMTHFの結合に関与する His44は,ラン色細菌ではAla45となっており,8-HDFが結合するための空間を与えて いる.Phe80も大腸菌では8-HDF結合サイトを塞いでいるが,ラン色細菌ではこの付 近のアミノ酸が欠損している.Gly106は8-HDF結合サイトのスペースを与えるのに ちょうど良いが,大腸菌ではMrHFとの結合に必須のアミノ酸であるGlu109に置き 換えられている.ラン色細菌のLys248も8-HDFと水素結合をする重要な残基である が,大腸菌ではAla242となっている.逆に大腸菌のLys293はMTHFとの水素結合に必 須であるがラン色細菌では欠損している.
大腸菌はその生活環内では8-HDFを生産することはできない.一方,ラン色 細菌は8-HDFを持つことによって,より長渡長側の光を集光することができた.分 子進化的に考えると,ラン色細菌がより長波長の光を獲得するために本来MrHFが 結合していた部分のアミノ酸を少しずつ変異させて進化し,8-HDFの結合サイトを 持つことができたものと思われる.
ラン色細菌由来の光回復酵素の結晶構造を大腸菌由来の酵素との構造を比較 することによって,両タンパク質の集光補欠分子の認識機構を理解することができ た.非常によく保存された一次構造と同一のフォールディングを持った2つのタン パク質において,その結合部分のアミノ酸残基を変異させて種類の異なる補欠分子 を保持している例は,今までに確認されたことがなく,構造生物学上非常に興味深 いと思われる.
5.おわりに
本研究は東北大学加齢医学研究所安井明教授,オランダ,ロッテルダム・ エラスムス大学のA.P.M. Eker博士との共同研究で,京都大学大学院理学研究科化 学専攻の生物構造化学研究室(東京工業大学資源化学研究所生物資源部門からの継 続)において行われたものである.
この研究を行うにあたりX線回折実験で大きなご助力をいただいた高エネル ギー加速器研究機構,物質構造科学研究所(放射光研究施設),坂部知平名誉教授, 中川敦史博士(現北大院理),渡邊信久博士,鈴木守博士に御礼申し上げたい.
参考文献