1 偼偠傔偵 僼儔價儞娨尦峺慺偼揹巕嫙梌懱偲偟偰NADH傑偨偼NADPH傪梡偄丄FMN側偳偺僼儔 價儞椶傪娨尦偡傞峺慺偱偁傝丄嵶嬠偐傜傎擕椶傑偱峀偔暘晍偟偰偄傞丅摿偵丄敪岝嵶嬠 偵偍偄偰僼儔價儞娨尦峺慺偺惗惉暔偱偁傞FMNH2偑丄敪岝峺慺儖僔僼僃儔乕僛偺婎幙偵側傞偲峫偊傜傟丄拲栚偝傟偰偒偨丅
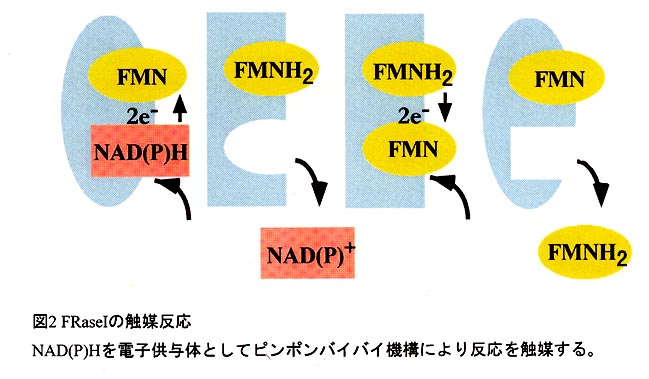
敪岝嵶嬠偵偍偗傞敪岝斀墳偼丄尰嵼偺偲偙傠丄埲壓偺傛偆側僒僀僋儖偱恑峴偡傞偲 峫偊傜傟偰偄傞1)丅偡側傢偪丄NAD(P)H:FMN oxidoreductase偱娨尦偝傟偨FMNH2偑儖僔僼僃儔乕僛偲寢崌偟丄偙傟偑峺慺偲斀墳偡傞偲丄拞娫懱偱偁傞C(4a)-hydroperoxy乚FMN偑惗惉偝傟傞丅偙偺僼儔價儞拞娫懱偲傾儖僨僸僪偑斀墳偟偰C(4a)-hydroxy-FMN偑偱偒丄偙傟偑婎掙忬懺偵栠傞嵺偵岝偑曻幩偝傟傞丄偲偄偆傕偺偱偁傞丅
(1)NAD(P)H亄FMN亄H亄仺NAD(P);亄FMNH2丂僼儔價儞娨尦峺慺
(2)FMNH2亄R-CHO亄02仺R-COOH亄H20亄h兯丂儖僔僼僃儔乕僛
敪岝嵶嬠Vibrio fischeri偺僼儔價儞娨尦峺慺FRaseI偼偦偺僐乕僪偡傞堚揱巕偐傜梊憐偝傟傞傾儈僲巁攝楍偑丄戝挵嬠僯僩儘娨尦峺慺NfsB2,3)傗岲擬惈嵶嬠Thermus themophilus HB8偺NADH巁壔峺慺NOX4)偲憡摨惈傪帩偭偰偄偨丅偟偐偟摨偠Vibrio懏偱NADPH摿堎揑偵摨條偺斀墳傪怗攠偡傞Vibrio harveyi偺僼儔價儞娨尦峺慺FRP5)偲偼桳堄側憡摨惈偼尒傜傟側偐偭偨丅
2 FRaseI偺X慄寢徎峔憿夝愅
奀梞惈偺敪岝嵶嬠Vibrio fischeri偺僼儔價儞娨尦峺慺FRaseI偺寢徎偼丄PEG4000傪捑揳嵻偲偟偰僗僩儕乕僋僔乕僨傿儞僌傪梡偄偨忲婥奼嶶朄偵傛傝摼傞偙偲偑偱偒偨6)丅摼傜傟偨寢徎偼嬻娫孮C2丄奿巕掕悢a=101.6侌丄b=63.3侌丄c=74.4侌丄兝=100.0亱偺扨幬徎宯偱丄旕懳徧扨埵偵儌僲儅乕2暘巕偑娷傑傟偰偄偨丅夞愜嫮搙僨乕僞偺廂廤偼幚尡幒宯偺R-AXlS 嘦c偲PF偺嶁晹儚僀僙儞儀儖僌僇儊儔傪暪梡偟偰峴偭偨丅埵憡偼廳尨巕摨宆抲姺 朄傪梡偄偰寛掕偟偨丅
儌僨儖偺峔抸偼僾儘僌儔儉O傪梡偄偰峴偄丄暘夝擻10.0乣1.8侌偺僨乕僞偵懳偟偰X乚PLOR傪梡偄偰惛枾壔傪峴偭偨丅嵟廔揑側儌僨儖偼N枛抂儊僠僆僯儞傪彍偄偨217巆婎偐傜峔惉偝傟丄R場巕=18.7%丄free場巕=21.3%偱偁偭偨丅儁僾僠僪寢崌偺棟憐抣偐傜偺偢傟傪昞偡RMS偼丄寢崌挿偱0.011侌丄寢崌妏偱1.62亱偱偁偭偨丅
3 FRaseI偺棫懱峔憿7)

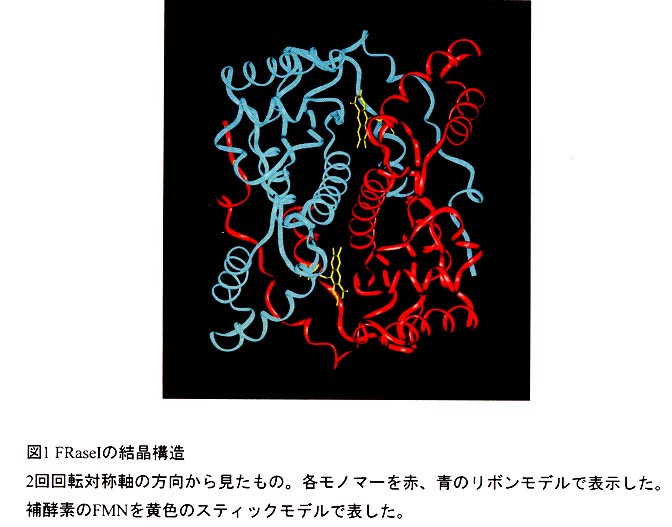
恾1偵FRaseI偺棫懱峔憿傪帵偡.杮峺慺偼妶惈拞怱偵曗峺慺FMN傪傕偮僼儔價儞僞儞僷僋幙偱偁傝丄9杮偺兛傊儕僢僋僗偲5杮偺僗僩儔儞僪偐傜側傞兝僔乕僩偱峔惉偝傟傞奺儌僲儅乕偑丄屳偄偵枾愙偵憡屳嶌梡偟偨儂儌僟僀儅乕偱偁傞丅曗峺慺偺FMN偼2偮偺儌僲儅乕偺奅柺偵懚嵼偟偰偍傝丄億働僢僩偺墱怺偔偵杽傕傟偰偄傞丅曗峺慺FMN偲峺慺偲偼嫟桳寢崌偱偼側偔丄廃曈巆婎偲10杮傪挻偊傞悈慺寢崌傪宍惉偡傞偙偲偵傛偭偰偒傢傔偰埨掕偵寢崌偟偰偄傞丅偙偺僰僋儗僆僠僪寢崌條幃偼丄僰僋儗僆僠僪寢崌峔憿偲偟偰戙昞揑側儘僗儅儞僼僅乕儖僪偲偼堎側傞傕偺偱偁傞丅杮峺慺偼丄戝偒偔寴偄僐傾傪宍惉偡傞僪儊僀儞偲丄僐傾偐傜撍偒弌偨2杮偺兛傊儕僢僋僗偐傜側傞僼儗僉僔僾儖側彫偝側僪儊僀儞偐傜峔惉偝傟傞丅偙偺峔憿傪尒傞偲妶惈拞怱偱偁傞曗峺慺FMN偲僼儗僉僔僽儖側僪儊僀儞偵偼偝傑傟偨椞堟偵婎幙偑寢崌偡傞偲峫偊傜傟傞丅偟偐偟丄偦偺億働僢僩偺戝偒偝偐傜峫偊偰婎幙偱偁傞NADH偲FMN偑摨帪偵寢崌偡傞偙偲偼偱偒側偄偟丄曗峺慺FMN偺棤懁偼姰慡偵僞儞僷僋幙偺巆婎偱暍傢傟偰偄傞偺偱丄2偮偺婎幙偑摨帪偵曗峺慺FMN偵寢崌偡傞偙偲偼晄壜擻偱偁傞.偙偺偙偲偼丄杮峺慺偺怗攠斀墳偑僺儞億儞僶僀僶僀婡峔偵廬偆偙偲偲堦抳偟偰偄傞(恾2)丅
4 慾奞
杮峺慺偺僼儔價儞娨尦妶惈偼僋儅儕儞桿摫懱偵傛偭偰慾奞偝傟傞偙偲偑尒弌偝傟丄偦偺慾奞婡峔傪峺慺斀墳懍搙榑揑偵夝愅偟偨寢壥丄偙傟傜偺慾奞嵻偼戞堦婎幙偱偁傞NAD(P)H偵懳偟偰嫞崌揑偵丄傑偨戞擇婎幙偱偁傞FMN偵懳偟偰偼晄嫞崌揑偵嶌梡偡傞偙偲偑敾柧偟偨8)丅傑偨慾奞掕悢偼KI偲偟偰10-7乣10-5M偱丄旕忢偵嫮椡偵杮峺慺偺僼儔價儞娨尦妶惈傪慾奞偡傞偙偲偑傢偐偭偨丅偁傞婎幙偵懳偡傞嫞憟慾奞嵻偼丄偦偺婎幙偲摨偠晹埵偵寢崌偟丄峺慺堦慾奞嵻暋崌懱(EI暋崌懱)傪宍惉偡傞偙偲偵傛偭偰妶惈傪慾奞偡傞偺偱丄峺慺堦慾奞嵻暋崌懱偺峔憿傪夝柧偡傟偽丄婎幙寢崌晹埵傪摿掕偡傞偙偲偑壜擻偵側傞丅偦偙偱僜乕僉儞僌朄偵傛傝暋崌懱偺寢徎傪嶌惉偟丄暋崌懱偺寢徎峔憿傪寛掕偟偨丅
5 暋崌懱寢徎峔憿
dicoumarol暋崌懱偺峔憿傪恾3偵帵偡7).偙傟傪尒傞偲慡懱偲偟偰偼愭掱偺峺慺偩偗偺巁壔宆峔憿偲戝偒側曄壔偼側偄.偟偐偟曗峺慺FMN偺嬤朤傪奼戝偡傞偲丄妶惈晹埵偱偁傞偲峫偊偰偄偨椞堟偵dicoumarol偑寢崌偟偰偄傞.dicoumarol偼丄Ie43偺傾儈僪拏慺偲悈慺寢崌傪宍惉偟偰偍傝丄傑偨曗峺慺FMN偺僀僜傾儘僉僒僕儞娐偲Phe124懁嵔偺朏崄娐偲偺娫偵嫴傑傟偄偨.偙偺椞堟偼敿宎栺4侌丄挿偝栺12侌偺墌摏宍偺僒僽億働僢僩偲側偭偰偍傝丄媡懁偺梟攠椞堟偵撍偒敳偗偰偄偨丅
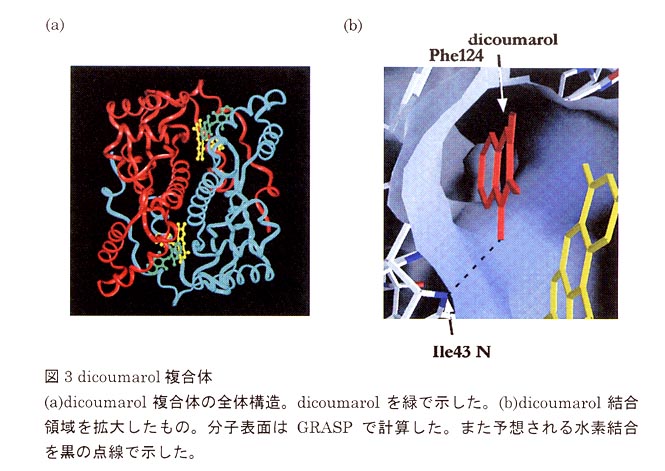
傑偨dicoumarol埲奜偺慾奞嵻傕慡偰Ile43傾儈僪拏慺偲悈慺寢崌傪宍惉偟偰偄偨8)丅偙偺悈慺寢崌傪宍惉偱偒側偄僋儅儕儞偑丄杮峺慺偵懳偡傞慾奞嵻偵側傝摼側偐偭偨偙偲傪峫偊傞偲丄偙偺1杮偺悈慺寢崌偑儕僈儞僪寢崌偵昁恵偱偁傞偙偲偑帵嵈偝傟傞丅偙傟傜慾奞嵻偑NAD(P)H偺嫞憟慾奞嵻偱偁傞偙偲偲丄僒僽億働僣僩偺戝偒偝傪峫椂偡傞偲丄NAD(P)H偺巁壔娨尦晹埵偱偁傞僯僐僠儞娐偑偙偺椞堟偵寢崌偡傞傕偺偲峫偊傜傟傞丅
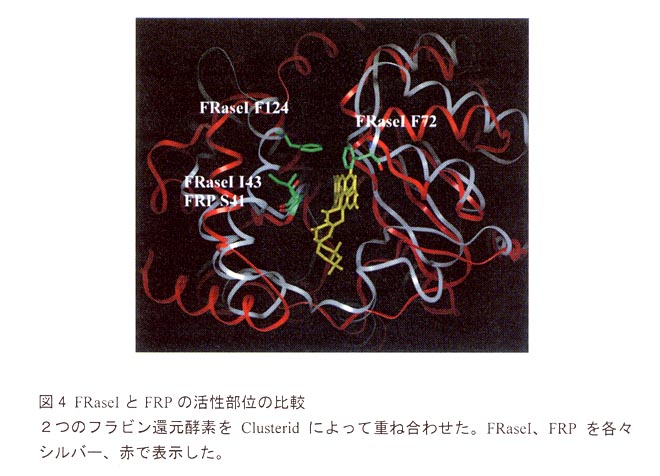
Vibrio harveyi偺僼儔價儞娨尦峺慺FRP傪廳偹偁傢偣傞偲丄FRaseI偺ne43偵偼FRP偱偼Ser41偑憡摉偟偨(恾4)丅偟偐偟曗峺慺FMN偵懳柺偡傞僿儕僢僋僗偺憡懳埵抲偑堎側偭偰偄傞偨傔丄FRP偵偼FRaseI偺Phe124偵懳墳偡傞巆婎偑側偄丅埲忋偺寢壥傛傝丄2偮偺僼儔價儞娨尦峺慺偱偼丄僺儕僕儞僰僋儗僆僠僪偺寢崌條幃偑堎側傞偙偲偑帵嵈偝傟傞丅
6 偍傢傝偵
埲忋偺傛偆偵丄慾奞嵻暋崌懱傪峫椂偡傞偙偲偵傛偭偰丄懠偺僼儔價儞娨尦峺慺偲偺婎幙摿堎惈偺憡堘傪帵嵈偡傞偙偲偑偱偒偨丅崱屻偝傜偵丄敪岝峺慺儖僔僼僃儔乕僛偲偺僞儞僷僋幙娫憡屳嶌梡傪夝柧偟偰偄偒偨偄丅
杮尋媶偼搶嫗戝妛惗暔惗嶻岺妛尋媶僙儞僞乕彫抮塸柧攷巑(尰岺嬈媄弍堾惗柦岺妛岺嬈媄弍尋媶強)丄嵅乆栘岹彆庤丄搶嫗戝妛戝妛堾棟妛宯尋媶壢惣嫿棾嫵庼丄慞栰廋暯攷巑丄儚僔儞僩儞戝妛E.T. Adman嫵庼丄僽儕僥傿僢僔儐僐儘儞價傾戝妛M.E.P. Murphy攷巑偲偺嫟摨尋媶偱偁傞丅
杮尋媶傪悑峴偡傞偵偁偨傝丄X慄夞愜幚尡偱屼彆椡捀偄偨崅僄僱儖僊乕壛懍婍尋媶婡峔嶁晹抦暯柤梍嫵庼丄搉曈攷巑丄楅栘攷巑丄屲廫棐攷巑偵怺偔姶幱偄偨偟傑偡丅
嶲峫暥專