
はじめに
生体高分子のX線結晶解析のペースは飛躍的に改善されてきている。シンクロトロン放射光の果たしてきた役割は言うまでもなく、コンピュータの格段の進歩、優れたソフトウエアの開発、遺伝子工学の長足の進歩など様々な要因が考えれる。しかし今でも時間のかかる段階は結晶化であり位相付けの段階であろう。このため構造解析はやはり時問のかかるものとなっている。ここをいかにしてクリアするか?
その一つの手段として筆者らが行っている融合蛋白質を用いた結晶化を紹介し、有利な点と問題点をあげてみたい。
1-1 サンプル調整−アフィニティタグの利用
興味のある蛋白質の構造解析を行う場合、大腸菌を利用した大量発現系を用いるのが一般的である。この段階の全般的な話は種々のプロトコール本(1)やベクター、コドンの選び方は文献(2)があるのでそちらを参照されたい。さらに精製の容易さを考慮してタグ付きの蛋白質一グルタチオン-S-トランスフェラーゼ(GST)やマルトース結合蛋白質(MBP)、さらにはHis-tag-として発現させることが多い。融合蛋白質として発現させると可溶成分にきやすいというメリソトもある。一方、タグ部分は"余計な"ものなので後に切り離せるようにトロンビンなどのプロテアーゼのサイトを入れてあるのが普通である。最近は一歩進んで、非特異的な切断を避けるため厳密な認識を行うとされているプロテアーゼを用いるPrecision system(ファルマシア)やProteinsplicingを利用したIMPACTシステム(バイオラボ)などもキットとして売り出されている。
1-2タグ付きのままの結晶化
タグ付きの蛋白質は確かに精製は容易になるが、プロテアーゼによる切り離しの際に問題が生じることがある。すなわち非特異的に切られてしまったり、あるいは非常に切れにくかったりする。それではこのまま結晶化させたら"余計な部分"が邪魔をして結晶が出ないのか?既にGSTの立体構造は解析されているので、もし結晶化に成功したなら構造解析は分子置換でうまくいくのではないか?このようなことを考えて行ったのが以下の研究である(3)。
1-3 GST-DREFの結晶化
ショウジョウバエの遺伝子複製に関わる蛋白質の多くは、そのプロモーター領域にDRE(DNAreplication-relatedelement)配列を有している。そこに特異的に結合して転写制御を行うのがDREF(DRE-binding factor)である(4)。DREFはそのN末領域にDNA結合ドメインを有している。我々はDREFによるDNA認識機構を原子レベルで明らかにするため、DNA結合活性を有しているDREF(16-105)(以下DREF(105)と略す)の構造研究に着手した。DREF(105)はGST融合蛋自質としてプラスミドが供与されていたため、当初はトロンビン処理によってGSTを切り離して結晶化条件を探索した。数多くの結晶化条件を試行したものの、X線結晶解析に適した結晶を得ることはできなかった。そこでGSTl)REF(105)のままで結晶化をすることにした。GST-DREF(105)のままでもDNA結合活性があることは確認済みである。ハンプトン社のスクリーンキットを用いて結晶化条件を探索したところ表lに示すような条件で結晶を得ることができた。これをもとに条件を精密化したところ、図1に示すような条件のとき良好な結晶を再現よく得ることができた。得られた結晶をよく洗浄し溶かし た後、SDS-pageを行うと予想位置(37KDa程度)のみにバンドが確認された。

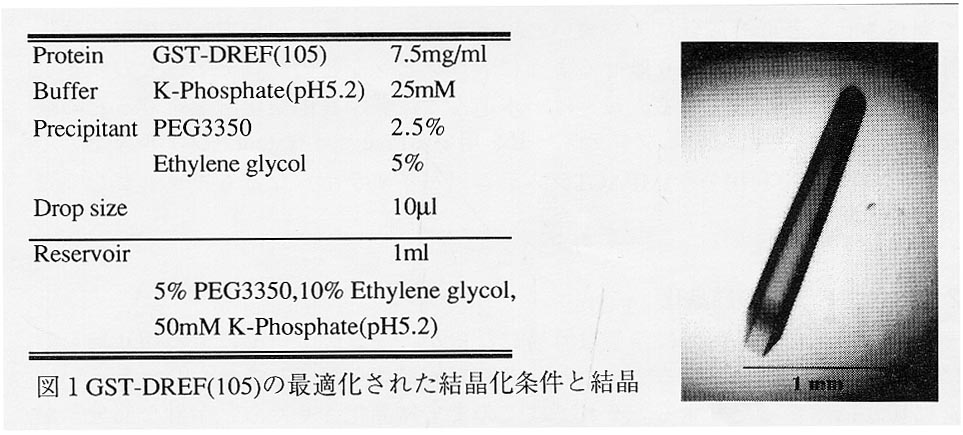
ここで我々が経験したことは、結晶化に用いるGST-DREF(105)はGlutathione sepharoseからの溶出サンプルを用いなければならない、ということである。最終カラムを、例えばイオン交換やゲル濾過にしてしまうと結晶が得られなくなった。これはGSTからグルタチオンがはずれてしまい結晶化条件が変わってきたり、あるいはGSTの不均一性(グルタチオンが結合しているものと結合していないものが混在)が生じるためだと思われる。その証拠に、結晶を与えなくなったサンプルでもGSHの添加により結晶を得られるようになった。しかしこの場合は再現性が劣り、結晶の質も悪かった。
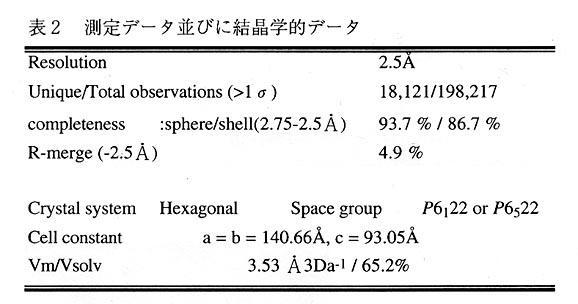
1-4 データ収集と分子置換
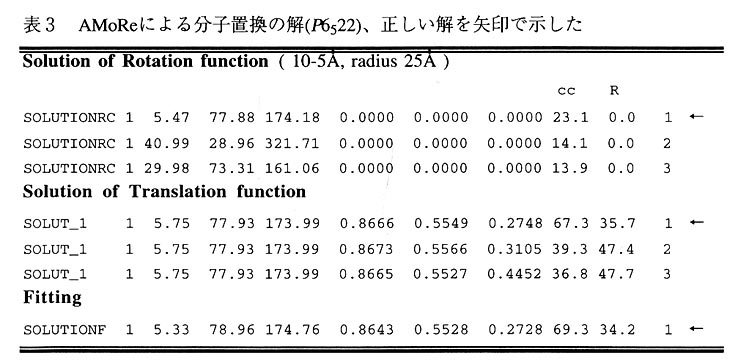
データ収集は(株)理学のX線発生装置FRと回折計R-Axis lVを用い、2.5Å分解能のデータを収集することができた。X線損傷を防ぐため-180℃の極低温中での測定である。表2に測定データと結晶学的データをまとめたものを示す。分子置換は既に構造解析されているSchistosoma japonicum(Sj)由来のGST(PDB code:1GNE(5)をモデルとしてプログラムAMoRe(6)を用いて行ったところ、表3に示すように容易に解を得ることができた。ここまでは順調であったが、残念ながら末だにDREF部分の構造は決定できないでいる。
1-5 結晶化条件の類似性
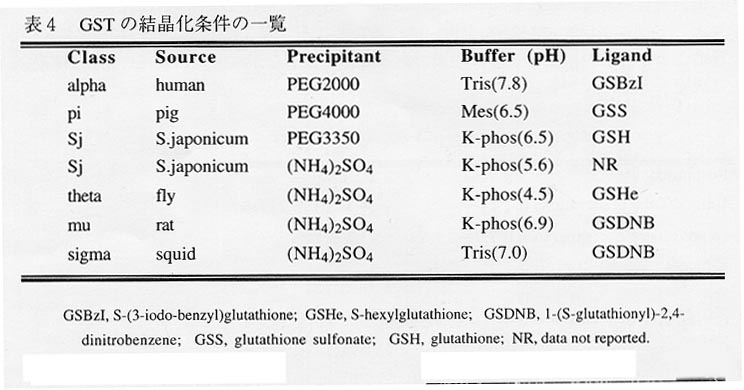
今回得られたGST-DREF(105)の結晶化条件は、表4にあげたような既に構造解析されたGST単独のそれと高い類似性を持っている。特に同じSj型を用いたLim et a1.(4)の条件とは高い類似性を示している。全体的にみても、沈殿剤としてはPEGあるいは硫安が用いられていることや、緩衝液として中性から弱酸性のリン酸バッファーが用いられていることが目に付く。GST-DREF(105)でも結晶が得られたのはすべてそのような条件の時であった(表1参照)。特に沈殿剤としてPEGが用いられていることは、後に述べるGSTを用いた融合蛋自質の結晶化にも多くの場合あてはまる。
1-6 他の蛋白への適用の可能性
図2結晶中でのパッキング

現在得られているGSTの結晶中でのパッキングの様子を図2に示す。斜め線は結晶格子で、トレース図がGST分子である。DREF(105)は自い円内に入り込んでいるはずである。それではこの中にどの位の分子量の蛋白質が入り込めるだろうか?このパッキングをとるとすれば直径約60Åの空洞があるので、直径約30Å程度の分子なら入り込めそうである。分子量でいうとせいぜい二万程度であろうか。しかしこの程度の分子量でも例えば機能ドメインの構造研究とすれば十分に有効範囲は広がるであろう。
1-7 融合蛋白質の結晶化一世界の情勢
融合蛋白質のまま結晶化させるという発想自体は特に新奇なものではない。恐らく最初の成功例は1994年に報告されたGSTとHlV由来のgp41の部分ペプチド(6残基)と思われる(5)。但しGSTとペプチドの問には9残基のリンカーが存在しているので、本来のGSTよりも15残基長いことになる。融合相手がペプチドではなく機能ドメインである場合も、我々の報告に引き続きいくつか発表されている。これらを表5にまとめた。このうち立体構造まで報告されているのはHlVgp41(5)とNa,K-ATPaseのAnkyrin結合ドメイン(7)という、どちらかと言えばペプチドレベルのものである。また、300残基を越える蛋白質も融合蛋自質として結晶化していることは注目に値する。

1-8 融合蛋白質における解析の間題点、解決法
先ほども述べたようにGST-DREF(105)の構造解析は成功していない。これには幾つかの原因が考えられる。
1) DREF(105)部分がしっかりした構造をとっていない
DNA結合蛋白質はDNAと結合していないとき、DNA結含領域が明確な構造をとっていないことがある。DREF(105)もDNA結合蛋白の一つであるのでその可能性はある。しかし、DREF(105)単独で一次元のNMRスペクトルをとると、ピークは十分に分離しており、部分的であればともかく、全体が構造を持たないとは考えにくい。この場合の解決法は、認識配列を含むDNAとの共結晶化が考えられる。
2) 結晶中における静的なディスオーダー
現在一番可能性があると考えている原因である。GST部分は結晶中で強くパッキングしている。このことは分子置換の解が容易に得られたことや、重原子を用いた同型置換法から得られたGST部分の電子密度が明瞭であることからも支持される。反面、DREF(105)部分の電子密度は不明瞭でトレースが不可能である。重原子サイトもGST部分にしか見当たらな い。このことから、それほど強くパッキングしておらず結晶中で静的にディスオーダーしていると考えられる。
この場合の一つの解決法はリンカーの長さであると考えている。GST-DREF(105)の場合は6残基のリンカーが存在する。さらにGSTのC末部分はかなり柔軟性に富んでおり、リンカーと相まって静的なディスオーダーを生み出しているものと思われる。図3にGSTとの融合蛋白質として解析に成功している構造の重ね合わせ図をのせる。GSTのC末部分の コンフォメーションが大きく異なることがわかる。Centerら(9)はMBPとの融合蛋白質の結晶化成功の原因はリンカーの長さであることを強調している。30残基にも及ぶリンカーを用いた場合は結晶がまったく得られなかったがこれをAla3残基にしたところ良好な結晶が得られた、というものである。
1-9 自動結晶化への展望
最終的には融合蛋白質を用いた自動結晶化であるが、まだ検討すべき点は多い。表5にあげた報告例のうち、蛋白レベルでの解析の成功例がまだ一つもないことも検討の余地があることを示唆している。まずは適用範囲というものを見極めなければならないであろう。分子量の制限、リンカーの検討、蛋白質同士の接触の仕方による制限など、課題はまだまだある。我々もGST-DREF(105)以外にも融合蛋白質の結晶化を行っており、別の融合蛋白質の結晶化に成功している。立体構造の解析に成功し、早くこれらの課題の検討をしたいと思っている。
1-10 他のタグの利用
融合蛋白質と並んでHisタグもよく利用される。付けたままで結晶化される場合もあるだろうが、切り離していることが多いであろう。Hisタグを付けたままの結晶化に関しては、ハンプトンリサーチ社のホームページに幾つかの意見が載っている。興味のある方は参照されたい(http://www.hamptonresearch.com/Literature.html)。
本題とはずれるが、結晶化を有利にするものとして抗体の利用が考えられる。膜蛋白質に抗体を結合させて親水性をあげた例も報告されている(13)。まだ融合蛋白質ほどは一般的ではないが、抗体を用いて結晶化の困難だった蛋白質を結晶化し構造解析に成功した例(例えば14)が報告されてきており大いに注目される。
参考文献
最近MBP融合蛋白質の構造解析がKobeらによって報告された
(Proc Natl Acad Sci U S A 1999 96(8):4319-24)。
これは融合蛋白質としては最初の成功例であるといえる。
(この部分の追加;1999.10)