タンパク質の立休構造の書式としてはBrookhaven National LaboratoryのProtein Data Bank(PDB)に登録される形式(PDB形式)のテキストファイルが一般的に用いられている。タンパク質は原子数が多く複雑なため、従来これをグラフィックスとして表示するには性能のよいコンピュータが必要とされていた。このためグラフィックス表示ソフトはその多くがUNlX上で開発されてきた。また、タンパク質の構造を表示する際には、全体をリボンで、注目するべき残基、リガンドをボールアンドスティックなどで描いた上に、水素結合や残基名のラベルなどを表示するのが一般的であるが、これはかなり複雑な作業であって、洗練されたインターフェースが必要となる。
現在はパーソナルコンピュータ(Windows、Mac)もUNlXと遜色ないものが比較的安価に入手できるようになり、またWindows、Mac用の構造ビューアもそれなりlこ使えるものが手に入るようになってきているが、今までに多くの人が使い、改良されてきたUNlXのプログラムとくらべると見劣りがするのが現状である。
ここでは現在よく用いられているUNlXベースの構造ビューアをいくつか紹介する。各ソフトウェアの図版はRibonuclease T1を同じ構図で表示するよう作成した。
1. RasMol
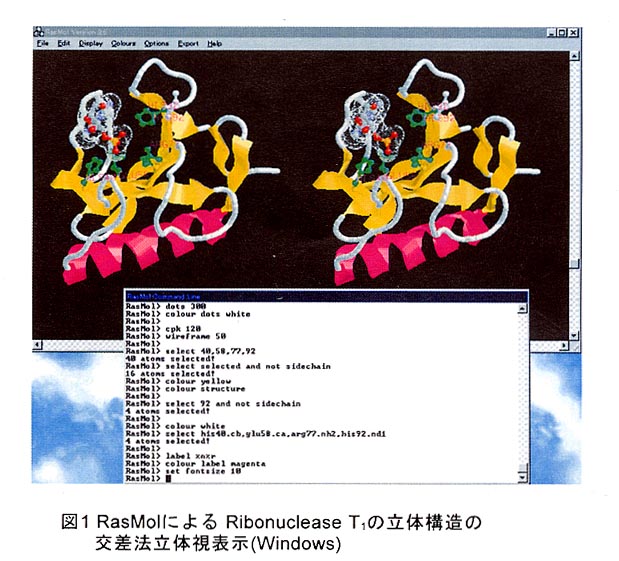
RasMoI1),2)はRoger Sayleによって開発されたソフトウェアで、Windows、MacおよびほとんどのUNlXに対応している。マウスを使ってインタラクティブに分子を動かすことのできるソフトウェアとしては極めて表示が速く、広く普及している。ただし、lnsight IIなどの商用ソフトや図の作成を目的としたソフトであるMOLSCRlPT、Raster3Dなどlこくらべるとグラフィックのクオリティは低めである(図1)。大まかな操作はプルダウンメニューから行い、ファイルのオープン、分子全体の描画方法の指定、画像ファイルの保存などができる。細かい描画方法の指定はコマンドラインから行う。例えばselectやrestrictのコマンドで原子を指定し、cpk、ribbonなどで表示方法を設定するというスタイルである。ワイヤーフレーム、ボールアンドスティック、スペースフィリング、リボンや二次構造毎にわかりやすく表示するなど、一通りのことができる。
残念ながら複数のファイルから分子を読み込んで重ねて表示することはできないが、カリフォルニア大学バークレー校のMultiCHEM facilityで開発されたUCB-RasMolという機能拡張版ではこれができるようになっている。また、RasMoIは次のMOLSCRlPTの形式でファイルを保存することができる。RasMoIはホームページ(表1)からダウンロードすることができる。


2. MOLSCRIPT
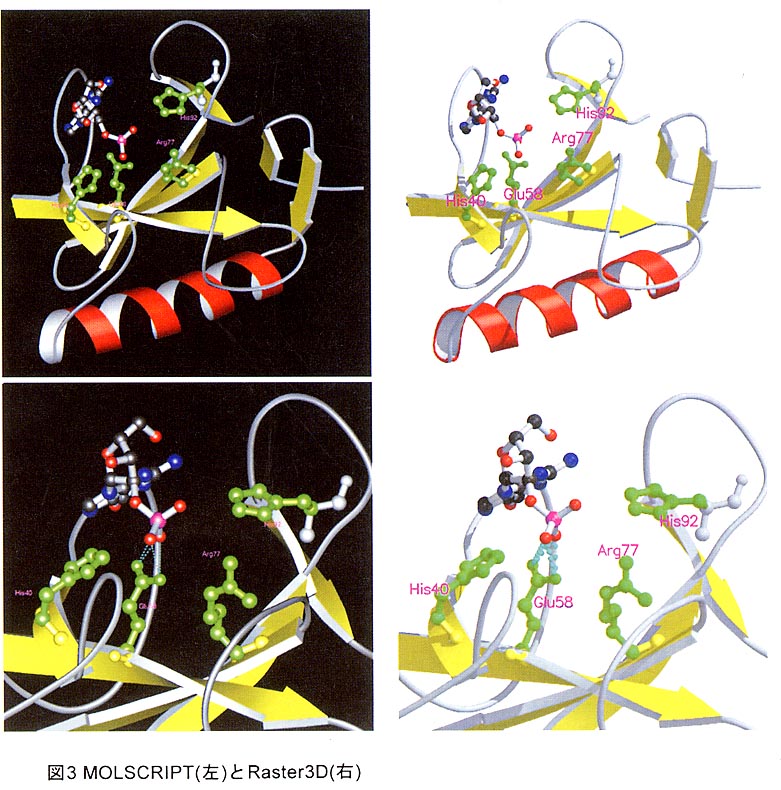
MOLSCRIPT3)は模式的な図を作ることができるブログラムである。ホームページでサポートされているのはSGI IRlX6.3だけだが、よく尋ねられる質問をまとめたぺ一ジ(Frequently Asked Question,FAQ)にlinuxとDEC Alphaについての記述があり、またほかのUNlXでも動作する筈だと書かれている。このプログラムで作成した図はモノクロでもわかりやすいというメリットがあり、論文などでよく使われている(図2)。また、別途グラフィックスライブラリOpenGLおよびGLUTを用意すればグラフィカルな図を作ることもできる(図3左)。SGIのマシンではOpenGLがはじめから入っているので、GLUTを表1のホームページから入手すればよい。GLUTがない場合でも次に述べるRaster3Dを使えば同様の図(図3右)を作ることができる。
このプログラムには図の描きかたを指定したテキスト形式の入カファイルが必要で、UNlXのプロンブトから入カファイルとPDBファイルの名前などを指定してMOLSCRIPTを実行すると画像ファイルが作られる(図2)。残基や原子ごとにCPK、スティック表示やリボン、ラベル、およびその色などを指定するので、簡単な図でも入カファイルは50から100行程度になる。OpenGLとGLUTのある環境ではインタラクティブに分子を動かして分子を見る方向を決めることができるビューアで確認しながら入力ファイルを作ることができ、作業がかなり楽になる。
入力ファイルをはじめから書くのは大変だが、分子のリボン表示やリガンドのボールアンドスティック表示など、大まかな描画の指定をして入カファイルを作ることができるMolAutoというプログラムがあるので、これを元に残基やテキスト、色の指定を書き足して描画させればよい。あるいは、RasMolを使ってMOLSCRIPT形式の入カファイルを作成することもできる(RasMolのコマンドラインからwrite molscript (filename)とする)。RasMolの出カは白黒のリボンモデルのみになるが、分子を見る方向も入カファイルに書き込まれるのが便利である。また、MOLSCRIPTを使って次に述べるRaster3Dの入カファイルを作ることができる。
MOLSCRIPTはPer J. Kraulisによって作られたプログラムで、アカデミックユーザーはホームページ(表1)からダウンロードすることができる。コマーシヤルライセンスを取得するにはホームページにある書式をプリントアウトし、必要事項を書き込んで郵送した上でライセンス料$3800を支払う必要がある。
3. Raster3D
Raster3D4),5),6)はDavid J. BaconとWayne F. Andersonによって作られ、Mark Israel、Stephen Samuel、Michael Murphy、Albert Berghuis、Ethan A.Merrittらが改良を加えたプログラムで、多くのUNlXで動作する。また、Windowsでコンパイルする方法についても簡単な記述がホームページにある。
Raster3Dの最近の論文のタイトルにPhotorealistic MolecularGraphicsとあるとおり、このブログラムは模型として見てリアルな絵を作ることに長けている。特に影の入った絵が描けるのはおそらくRaster3Dだけであろう。また、XtalViewの電子密度表示をRaster3Dで読み込み、立体的な把握のしやすい図を作ることができる(Raster3Dのホームページにサンプル画像がある)。Raster3Dはいくつかのプログラムから成り立っており、ball、rod、ribbonのプログラムを使って入カファイルを作成し、renderを使って描画する。また、入カファイルはMOLSCRIPTで作成することもでき、MOLSCRIPTで作成したのと全く同じ構図の絵(図3右)が作成できる。MOLSCRIPTで作る絵がどちらかというと硬い質感を持つのに対し、Raster3Dではやわらかな雰囲気になる。筆者らはRaster3Dの絵は背景を白くした方がよいように思うが、これは好みの問題であろう。Raster3Dもホームページ(表1)からダウンロードすることができる。
4. GRASP
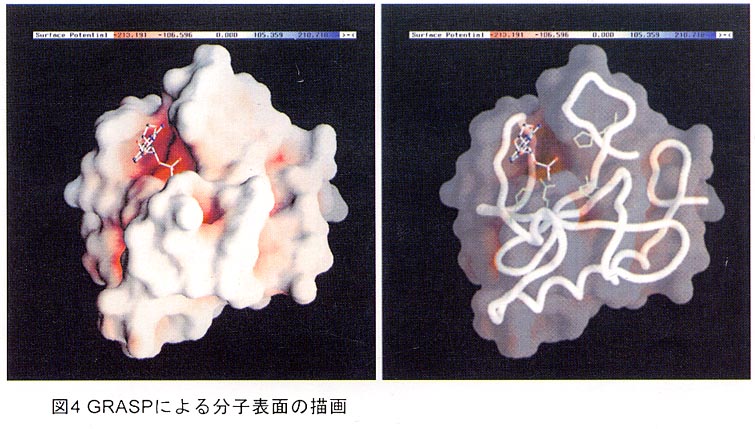
GRASP7)は静電ポテンシャルの計算と分子表面の描画ができるソフトウェアで、分子のかたちと電荷の分布を把握することができる(図4)。GRASPではマウスを使って分子を回転、移動することができ、また右クリックで出るメニユーでほとんどの操作ができるようになっている。コマンドラインも使え、さらにパネルという新しい操作方法が現在作られている。
GRASPは分子表面だけではなく、リボン、スティック、CPKなどの表示方法と組み合わせて使うことができ、また分子の切断面での静電ポテンシヤルの分布など、様々な解析、表現が可能である。
対応しているプラットフオームはSGIのみで、実行形式のファイルをホームページ(表1)からダウンロードするようになっている。GRASPは有償($500)なのでこのファイルはそのままでは使えない。ホームページのオーダーフオームに書き込んで送信するとGRASPを使えるようにする鍵が送られてくる。
以上見てきたような非営利のソフトウェアのホームページではインストール方法、マニユアルからTips、サンプル画像、関連ホームページヘのリンクなど参考になる情報がいろいろと提供されている。
5. Insight II
Insight IIはMSI社の製品で、高分子のモデリングを扱うソフトウェアとしては代表的なものである。Insight IIは単なるビューアというよりは、構造工ディタ、分子動力学計算、分子軌道計算、ホモロジーモデリング、リガンドデザイン、NMRおよぴX線による構造解析などを共通の環境で行うためのプラットフオームだと思われる。操作はマウスによるインタラクティプオペレーション+プルダウンメニューでわかりやすく、またマクロを組むこともできる(図5)。ここでとりあげた他のソフトウェアでできることはほとんどInsight IIでより簡単な操作でできるようになっている。
日本でのMSIの総代理店は帝人MSIであり、販売は菱化システム、CTCラボラトリーシステムズ、L.A.SYSTEMSなど8社が担当している(表1)。
なお、本稿でとりあげた以外にも様々な構造ビューアが発表されている。これらは、各ソフトウェアのぺージに必ずある、関連ソフトウェア、類似ソフトウェアヘのリンクページから探すことができる。また、詳しい説明などはないが、多数のソフトウ工アを表にしたものがNlHのCenter For Molecular Modeling(CMM)のホームページで利用できる。
表1 構造表示ソフトウェアのホームページ
1)Sanchez-Ferrer, A., Nunez-Delicado, E.,
Bru, R., (1995). Software for viewing biomolecules
in three dimensions on the Internet. Trends Biochem. Sci., 20, 286-288.
2)Sayle, R. A., Milner-White E. J., (1995).
RasMol: Biomolecular graphics for all. Trends Biochem. Sci., 20, 374-376.
3)Kraulis, P. J., (1991). MOLSCRIPT: a program
to produce both detailed and schematic plots
of protein structures. J. Appl. Cryst., 24, 946
4)Bacon, D. J. & Anderson, W. F., (1988).
A Fast Algorithm for Rendering Space-Filling
Molecule Pictures. J. Molecular Graphics, 6, 219-220.
5)Merritt, E. A. & Murphy, M. E. P.,
(1994). Raster3D Version 2.0: A Program for
Photorealistic Molecular Graphics. Acta Cryst. D, 5O, 869-873.
6)Merritt, E. A. & Bacon, D. J., (1997).
Raster3D: Photorealistic Molecular Graphics.
Methods in Enzymology, 277, 505-524.
7)Nicholls, A., Sharp, K., Honig, B., (1991).
Protein folding and association: insights
from the interfacial and thermodynamic properties
of hydrocarbons. Proteins, 11, 281-296.