はじめに
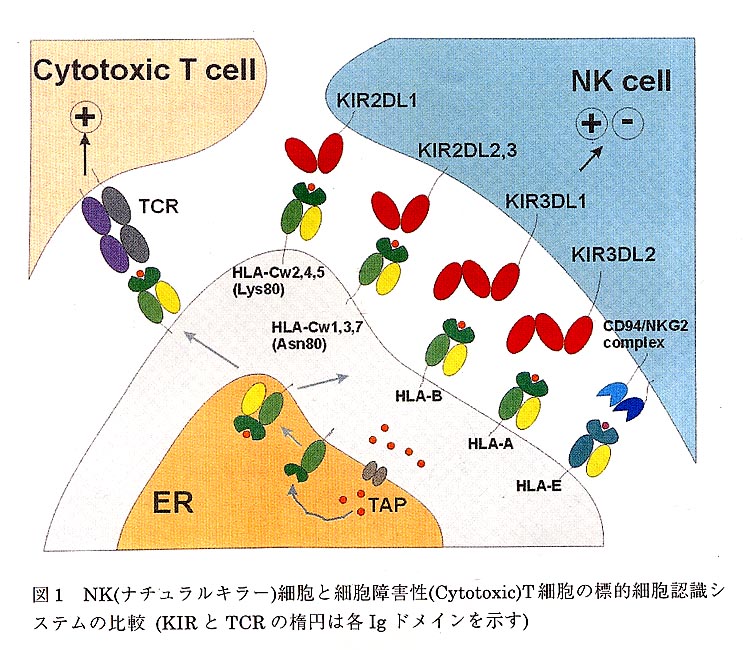
免疫系には初期の感染防御機構である自然免疫と抗原特異的な獲得免疫の2種類のシステムがある。獲得免疫を担うリンパ球の1つである細胞障害性T細胞では、有核細胞表面の主要組織適合性抗原複合体(MHC)クラスI(ヒトではヒト白血球抗原、HLA-A、HlMA-B、HLA-Cと呼ばれる)に結合しているウイルスや腫瘍由来の非自己ペプチドを抗原特異的T細胞レセプター(TCR)が認識し、正のシグナルを送り、MHC拘束性細胞障害活性を示す(図1)。他方、自然免疫に関わる同じくリンパ球で大型の顆粒をもつナチュラルキラー(natural killer、NK)細胞はウイルス感染細胞や腫瘍細胞などを抗原特異的刺激なしに殺したり、様々なサイトカインを分泌する。従来、NK細胞の細胞障害活性はMHC非拘束性であると考えられてきたが、最近いくつかのMHCクラスI分子に特異的に結合するNK細胞レセプター分子群が発見され、T細胞と同じくMHC拘束性を示すことがわかってきた。これらのNK細胞レセプターは、T細胞レセプターとは対照的にMHCクラスIを認識して負のシグナルを送り、結果として正常にMHCクラスI分子を発現している細胞は殺さない。逆に、MHCクラスI分子を発現していない異常な細胞はT細胞には認識されないが、NK細胞により殺される。T細胞とNK細胞はこの点において相補的役割を担っていると思われる(図1)。ヒトではMHCクラスI特異的なNK細胞レセプター群が2種類あり、1つはC-typeレクチンファミリーに属するCD941NKG2ヘテロ複合体ファミリーであり、他方は免疫グロブリン(Ig)ファミリーに属するヒトKiler cell immunog1obulin (Ig)-like receptor(KIR、Killer cell inhibitory receptor)ファミリーとヒトIg-like transcript(ILT)/leukocyto Ig-like receptor(LIR)ファミリーである。CD94/NKG2複合体はnon-classical MHCであるHLA-Eを認識するのに対して、KIRやILTはMHCクラスIを認識する(一部のILTはnon-classical MHCであるHLA-Gも認識するようである)。いずれのファミリーとも興味深いことにimmunoreceptor tyrosine based inhibitory motif(ITIM〉の存在する長い細胞内ドメインをもつものとITIMを持たない短い細胞内ドメインのものがある。短い方はimmunoreceptor tyrosine-based activating motif(ITAM)をもつDAP12などに結合する。それゆえ、このレセプター群はNK細胞の活性化とその阻害の両方に関与し、そのバランスでNK細胞の機能制御を行っていると考えられる1。
KIRは2つまたは3つの免疫グロブリン様ドメインを細胞外ドメインとしてもつtype I membrane receptor(それぞれKIR2DおよびKIR3Dと呼ばれている)である。KIR2DはヒトMHCクラスIのHLA-Cを認識し、XIR3DはHLA-Aまたは-Bを認識する(図1)。

KIR2Dは、大まかに2種類に分けることができ、HLA-Cw2,-Cw4,-Cw5を含むAsn77-Lys80をもつHLA-Cを認識するKIR2DL1と、HLA-Cw1,-Cw3,-Cw7を含むSer77-Asn80をもつHLA-Cを認識するKIR2DL2と3である。今回はHLA-Cw1,-Cw3,-Cw7に特異的なKIR2DL3について我々が行った結晶構造解析の話を中心にKIRのMHCクラス I に対する認識機構とその構造について説明したい。
実験と考察
KIRの大量発現系の構築
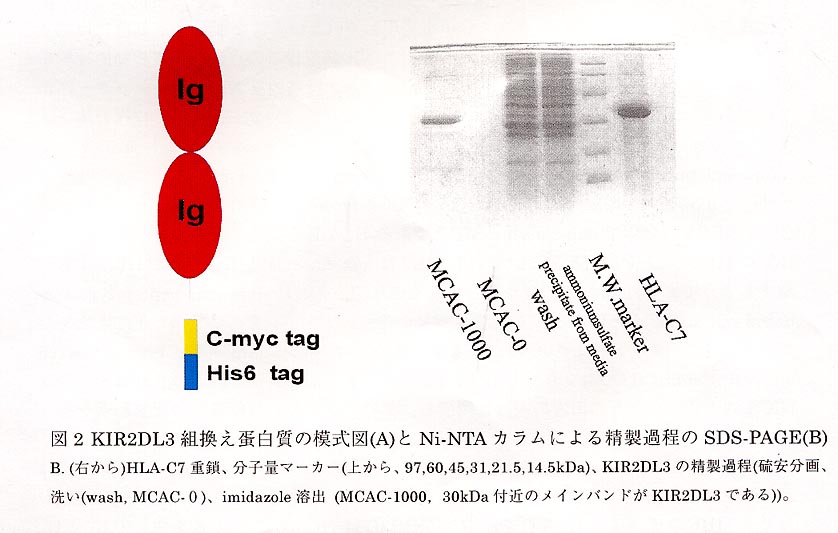
ヒト末梢血の白血球画分をIL2とPHAとともに培養し、得られた細胞からmRNAさらに cDNAを調製し、HLA-Cw1,-Cw3,-Cw7に特異的なKIR2DL3の細胞外ドメインの遺伝子を適当なプライマーを用いてPCR法により増幅した。これをNcoI及びNotI制限酵素サイトを用いて、p GEM2ベクター由来の分泌発現用ベクターp KMATHのT7プロモー夕ーの下流に組み込んだ。結果として、pelBシグナルをN末端に持ち、C末端にc-myc由来のリンカーとHis6-tagを持つ分泌発現ベクターを構築できた(図2左)。28℃で培養し、OD600=0.6-0.8で0.1mM IPTGにより発現誘導し、さらに一晩培養を続けた。組換え蛋白質は培養上清とペリプラズム画分に分泌され、これを硫安沈殿により回収した。さらにNi-NTAカラムとMonoQ(Pharmacia)カラムにより精製した(図2右)。最終収量は約0.5mg/lであった。
KIR2DL3の結晶化とX線データ測定
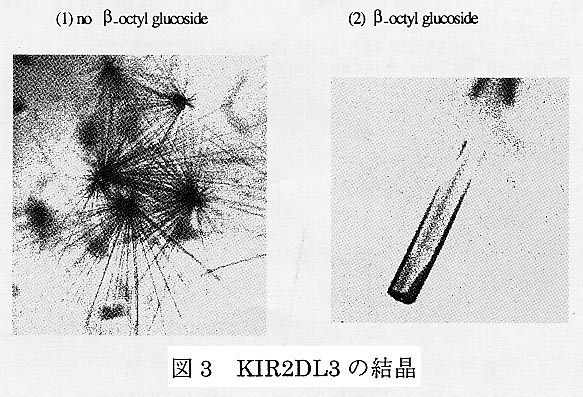
精製した蛋白質を5-10mg/ml濃度に濃縮し、結晶化条件をCrystal Screen(Hampton Research)で検討した。その結果、Crystal Screen II-37(10%PEG8000,0.1M HEPES pH7.5,8% Ethylene Glycol)で図3左のような針状結晶が得られた。そこで、pH、塩、有機溶媒などを加えて条件の改良を試みたが、うまく行かなかった。しかし、界面活性剤であるβ-octylglucosideを0.5%加えた時に棒状結晶が得られた2(図3右)。X線データ測定をPhoton FactoryのBL6Aで0.05x0.05x0.25mmの結晶を用いて15℃で行った。小さい棒状結晶ゆえに多少の異方性が見られたが、3Åまでのデータが得られた。指数付けとスケーリングをDenzoとScalepackで行った。
分子置換法による解析
すでにWileyらのグループが回じファミリーのKIR2DL1の結晶構造解析3を発表していたので、このモデルを用いて、分子置換法による解析を試みた。後程わかるようにKIRは2つのIgドメインからなり、これらの配置がKIR2DL1とKIR2DL3でかなり異なるため、分子全体では分子置換法による解析が成功しなかった。AmoreやX-PLORを用いて各ドメインでサーチしたが、うまく行かなかった。理由として考えられるのは、(1)まずデータの質があまりよくなく、かつ分解能が高くないこと、と(2)IgドメインはlP-sheetから成るため、裏表と上下の方向にstrandが伸びており、かつP-sheetの水素結合の流れがあるため解析を難しくしていると考えられた。そこでTongのCombined MolecularReplacementというプログラムを使った4。分子置換法では通常、計算スピードの節約のため、まずパターソン図をもとに分子の回転方位を決め、その後、並進量を決めていくという方法を行う。このプログラムはその回転関数の解の結果が通常それほどよくないことが多いことを踏まえて、どの範囲の回転関数の解でも並進の解析に当てはめることができるように作られている。また、このプログラムは分子の重なりをチェックすることができるので、多くの解をグラフィックス上で確認する作業を軽減することができる。Tongの論文中でも指摘されているようにパッキングのチェックがかなり有効であるようだ。我々のKIRの結果を図4に示す。

このように各ドメインで回転関数の上位40%の範囲の解を調べることにより、ピークがはっきりと得られた。また、この場合Fを用いずに規格化されたFを用いる方がよりよい結果を導いた。各ドメインの得られた解の位置関係が容易にドメイン問をつなぐことのできる位置にあり、正しい解が得られたと考えられた。Rigid-body Refinementを行い、その後、X-PLOR、Refhlac、CNSを用いて精密化を行い、Rcryst=24.8%(Rfree=32.0%)のモデルを構築できた5。
KIRの全体構造とドメイン構造
KIR2DL3の全体構造はKIR2DL1と似て、2つのIg-hkeドメイン(以下ではN末側のドメインをD1、C末側のドメインをD2と呼ぶことにする)がタンデムに並んだ構造をしており、他の細胞接着分子やレセプターに比べて、異常に急なelbow角を取っている(図5)。
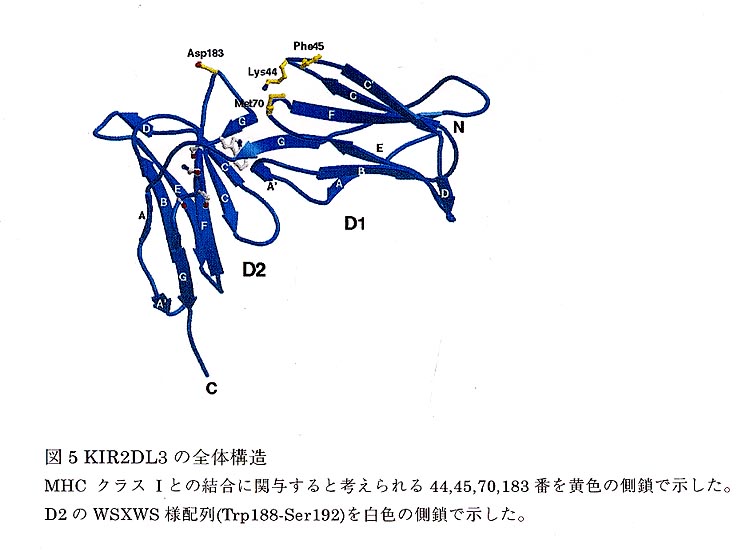
また、Sunらのグループが解析した、同じくHLA-Cw1,-Cw3,-Cw7に特異的なもう一つのメンバーKIR2DL26の構造も似ており、これはKIRファミリーの構造の一般的特徴と考えられた。各ドメインは旧来の分類分けではC2setに似ているのだが、A'strandを持ち、また、Dstrandのような構造も見られるため、厳密には新しいタイプのIg-hkeドメインだと考えられた。KIRドメインをstructural similarity search program Dali (version 2.1)で解析したところ、Hematopoietic Receptor(ヒト成長ホルモンレセプターやサイトカインレセプターが属している)に含まれるFibronectin type III(FN III)ドメインにも似ているが、より典型的なIgドメインに似ていた。またHematopoietic Receptorに特徴的に見られるTry-Ser-X-Serという配列のWSXWSボックスに類似したTrp-Ser-Asn-Ser-Ser(188-192番)という配列がKIRドメインにも見られた。しかし、Hematopoietic Receptorでは各Trpの芳香環の側鎖が同じドメイン内の他のストランドのArgやLysの側鎖と秩序だったパッキングをしているのに対して、KIRではTrp188がドメイン間の相互作用に関与しており、全く異なる構造を持っていた。
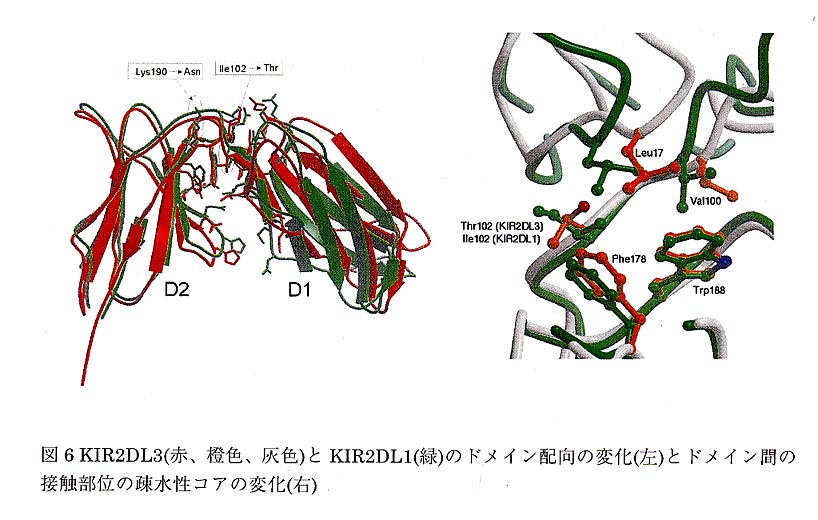
KIRドメインの配向の変化
当初、KIR2DL1の構造解析の結果から屈曲したこの構造はドメイン間の接触部位の広範な相互作用から来ており、この構造のFlexibilityは低いと考えられた。しかし、意外にもKIR2DL3の構造ではKIR2DLlと比べて、23度もドメインがずれていることがわかった(図6)。この為、KIR2DLlで見られたドメイン問のつなぎ目から遠いところの相互作用が見られず、この構造はつなぎ目に近い部分の相互作用に由来していると考えられた。他のKIR2DL2の二つの異なる結晶系の構造で5度ほどのドメイン間のずれが見られたが、急なelbow角を持つ構造は維持されていた。また、つなぎ目部分のドメイン問の相互作用に関わるアミノ酸をKIR2DL1,2,3で比較したところ、ほとんどのアミノ酸が維持されていたが、102番目のKIR2DL1ではne102が、KIR2DL2,3ではThrに変化していた。Thr102の側鎖の水酸基が存在しているため、周辺部のドメイン間のつなぎ目部分の疎水性コアの配置が変化していた(図6)。このことによりKIR2DL2,3の構造ではKIR2DL1よりも少しドメイン問のelbow角が緩やかなっているのかもしれない。このようなドメイン間の配向の変化がまたKIR2DL1とKIR2DL2,3のリガンドMHCクラスIの特異性に影響していることも考えられる。
MHCクラスI結合部位の構造
WinterとLong7はsite-directed mutagenesisによりKIR2D上のMHCクラスIの基質特異性を決定している部位が、D1のCC'ループの44番目の残基である事を同定した(図5)。44番目がLysであるKIR2DL2や3はHLA-Cw1,-Cw3, -Cw7を含むグループを認識し、他方MetであるKIR2DL1の場合はHLA-Cw2,-Cw4,-Cw5を含むグループに結合す る。この44番目残基を交換するだけで基質の特異性を変換することができる。また、他のグループの報告から、DlのCC’ループの45番、D1のEFループの70-72番やD2のFGループの183番も結合に重要な残基であることがわかった。他方、リガンドであるMHCクラスIのKIR2D結合サイトも同定されており、ペプチド結合部位のC末端側部分(73,76,80,90番)を認識する8。特に80番目の残基がLysである場合(HI」A-Cw4)、KIR2DL1を認識し、Asnの場合(HLA-Cw7)、KIR2DL2や3を認識する。この80番目残基を交換するだけでKIR2Dの特異性を変換することができる。
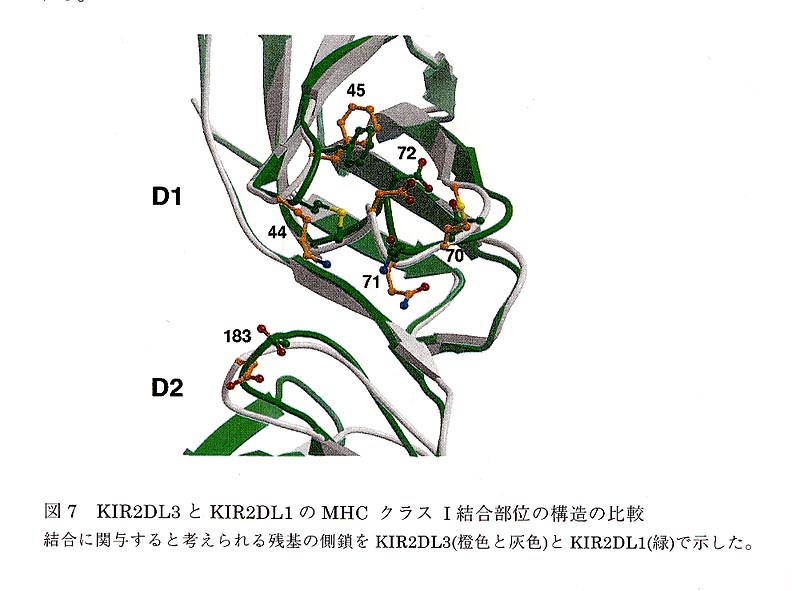
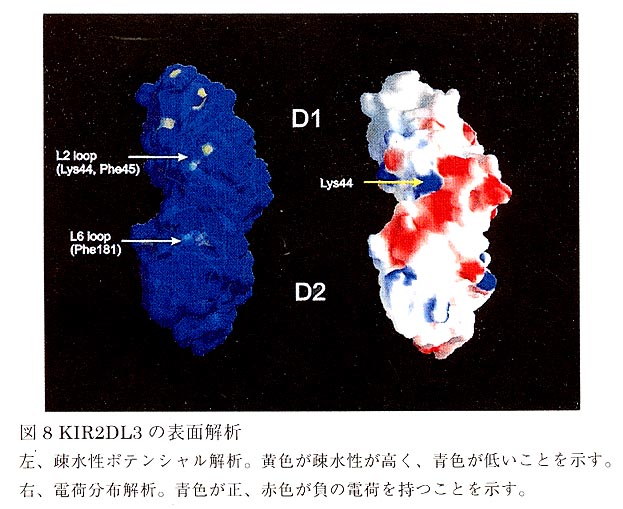
MHCクラスIの結合すると考えられる部位をKIR2DL1とKIR2DL3で比較したところ、上に上げた3つのループにはドメインの動きの影響が若干見られた(図7)。KIR2DL3のLys44(KIR2DLlではMet)になったことによる主鎖の大きな変化はほとんど見られなかったがJ」ys44およびPhe45の側鎖の配向がEFループの変化の影響を受けて、KIR2DL1のそれらとは異なっていた。KIR2DL3のPro68(KIR2DL1ではArg)のため、D1の疎水性コアの構成がKIR2DL1とは異なり、EFループの構造変化と同調しているようである。また、Lys44の側鎖NεがGln71と水素結合しているが、蛋白質の表面電荷の解析(図8右)から、Lys44の正電荷が表面に見られ、結合に影響することが考えられた。また、hydrophobic potential analysisの解析〈図8左)から、Lys44の疎水性部分が結合部位の中で強い疎水性を示した。KIR2DL1と2を用いた変異体の解析の結果はLys44の正電荷がLys80を持つHLML-Cw4の結合を妨げているようだが、単純に電荷だけで全てを説明できないので、疎水性部分とその立体構造も特異性および親和性を決定しているものと思われる。
KIR2DとMHCクラス I の相互作用の機能解析
我々はまたKIR2Dと数種のMHCクラスIを用いて、表面プラズマ共鳴を用いた結合実験(BIAcore)を行った(前仲ら、未発表)。その結果、KIR2DとMHCクラスIの結合の解離定数は数μMであり、この結合はHLA‐Cにアロ特異的であり、かつ結合しているぺプチドに依存していた。このような認識はT細胞レセプターのMHCに対する認識と似ているが、kineticsおよび熱力学的パラメーターが大変異なっていた。KIRのMHCに対する結合はkineticsがとても速く、T細胞レセプターの数十倍であり、他の細胞接着分子の相互作用と同じレベルであった。また、熱力学的に見ると、KIRの認識は、T細胞レセプターのような大きなエントロピーの損失を大きなエンタルピーで相補するような形ではなく、一般的な蛋白質−蛋白質相互作用にみられるものであった。
今後の展望
KIR/ILTレセプター群に属するメンバーの中にはNK細胞だけでなく、血球に幅広い発現分布を示すものがあり、これらの機構がNK細胞だけの特殊な例ではなく、広く一般的に免疫関連細胞にも及ぶようになってきた。これらのレセプター群の解析が進むことにより、血球全般の理解につながっていくと思われる。また、KIR/ILTファミリーの遺伝子群はヒト染色体19q13.4に存在しているが、Fcαレセプター(CD89)の遺伝子も同じ位置に存在しており、一次配列上の相同性も見られた。最近解析された他のFcレセプター(FcγレセプターIIbとFc8レセプター)の結晶構造9,10から、それらの2つのIgドメインの配向などはKIRと異なるが、elbow角が急である点やリガンド結合部位の位置がKIRと類似していることがわかった。これらのKIR/ILTファミリーとFcレセプターファミリーとの関連も興味深い。いずれのファミリーともレセプターとリガンドの複合体の解析はなされておらず、今後の課題となっている。また、細胞レベルでの機能に反映されているかもしれない、自然免疫に関わるKIRと獲得免疫に関わるT細胞レセプターのMHCクラスIに対する認識機構の違いも、これらの複合体の構造解析が進むことにより明らかになってくると期待される。
おわりに
KIR発現系の構築は日赤中央血液センターの十字猛夫所長、田所憲治副所長、屋部登志雄博士、中山貴博君、菊池安希子さんをはじめとする多くのセンターの方々と東京大学医学部徳永勝士教授の協力を得て確立することができた。KIRの結晶構造解析はオックスフォード大学E. Yvonne Jones博士とDavid I. Stuart教授との共同研究により行われ、機能解析はオックスフォード大学のAnton P. van der Merwe博士との共同研究で行われた。また、多くの助言をいただきました東北大学工学部熊谷泉教授、津本浩平博士、創薬技術研究所の松島正明博士、およびオックスフォード大学池水信二博士をはじめとするLaboratoryof Molecular Biophysicsの方々に感謝いたします。最後になりましたが、Photon Factory(PF)でのデータ測定におきまして大変お世話になりました坂部知平教授、渡辺信久博士をはじめ、PFの皆様に感謝いたします。
図3は文献2からIUCRの許可を得て転載しました。
図5,6右、7は文献5からElsevier Scienceの許可を得て転載しました。
文献 ( 解析の手法、装置、プログラムに関する文献は割愛させていただきました。)