はじめに 構造生物学的解析法(X線結晶解析,中性子線結晶解析,NMR,電子顕微鏡,各種プロー ブ顕微鏡など)が目覚ましい発展を遂げ,蛋白質を始めとする生体分子の立体構造解析が盛んになりつつある。もう一方で,ヒトを含めた多くの生物のゲノム解析が進行しつつある。これらの解析法と,長年の歴史をもつ,蛋白質工学を含めた蛋白賞機能解析法を基にして,「できることなら,約10万個の遺伝子からなるヒト個体全体について,蛋白質を含めた全生体分子の立体構造を解明し,それらの立体構造に基づいて分子機能を解析して,最終的にはヒトの全生命現象を,分子レベルだけではなく,原子レベルの分解能で理解したい。もしそれが可能になれば,医療などにも大いに役立つのだが,.....」という夢は,現時点の我々の学問レベルから考えると,当分の間,かなえられれそうにない。
ならば,「ヒトの遺伝子約10万個の中で,あらゆる生物に存在し,基本的生命現象に関与する重要な遺伝子約1000個を持つ1つの細胞についてだけでも,細胞全体の生命現象を,まず研究しておきたい。」それだけ重要な基本的遺伝子ならもう研究は終わっていてもよさそうだが,実は約113(約300個)の遺伝子は,全く機能未知である。もしその300個の遺伝子のうち,1つでも機能がわかれば,大発見である。
それでは,
(1)そのように重要な基本的遺伝子約1000個を持ち,
(2)生命現象を理解するために不可欠な"遺伝子操作系"と,
(3)立体構造解析や分子機能解析に必要な"耐熱性"とを,兼ね備えた生物はいるのだろうか?そのような視点で,この地球上に1億種類近く存在すると言われる生物を見廻してみると,高度好熱菌 Thermus thermophilus HB81)(図12))が最適であることがわかる。

この高度好熱菌は,今から約30年前に,日本の伊豆半島・峰温泉で,現東京薬科大学・大島泰郎教授によって発見された大きさ約5μmの真正細菌で,85℃の高温でも生育できる1)。
「構造生物学的解析や機能解析に適した本高度好熱菌細胞をモデル生物として選び,そこに存在する低分子・高分子すべての生体分子の機能を立体構造に基づいて解析して,『高度好熱菌 Thermus thermophilus HB8 丸ごと一匹』を原子レベルで理解すること」を最終目標とするのが,「高度好熱菌丸ごと一匹プロジェクト」である2)。これが進めば,1つの細胞全体の生命現象をシミユレーションするという夢へも近づく。
【1.細胞モデルとして高度好熱菌(T. thermophilus)を選んだ理由】
かつて,世界中の分子生物学者が,研究対象を大腸菌K‐12株,そのファージを入やT4,などと決めて皆で集中的に研究し,大成功を収めた。本プロジェクトの生物材料として高度好熱菌(T. thermophilus HB81)))(図1)を選ぶ理由は,
1)遺伝子操作系が確立した生物の中で,もっとも高温で棲息する。遺伝子操作に必要な薬剤耐性遺伝子(カナマイシン耐性遺伝子)にういては,DNAシャッフリング法を利用して耐熱化し,85℃でも使用可能な遺伝子を作製した3)。
2)蛋白質の安定性が高く,結晶化も容易なので,X線結晶解析・NMR等の構造生物学的
解析や機能解析に適している。
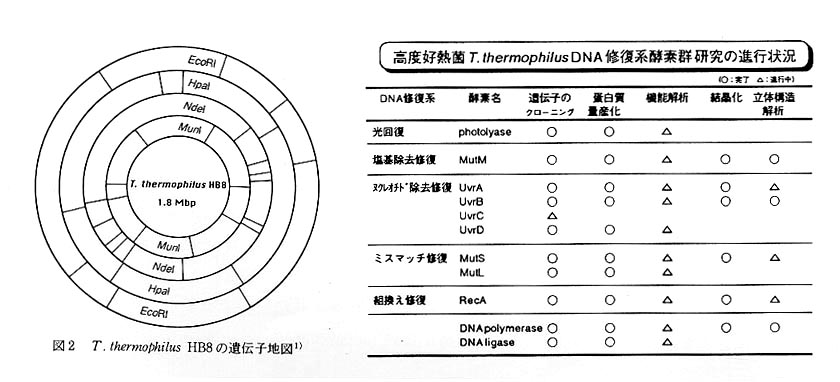
3)ゲノムサイズは1.8Mbp(図22))と小さく,細胞の生命活動に必須な遺伝子のみを進化の過程で保持してきたと考えられるが,最少培地で生育するために必要な遺伝子は一揃持っている。
4)大腸菌と共通点が多いため,大腸菌で確立された遺伝子操作系も使用可能である。
5)細胞にとって基本的で重要な酵素蛋白質(例えば,我々の研究室で解析を進めているDNA修復系酵素群など4,5)(右下の表))は,ヒトなどの高等動物とも相同性が高く,その立体構造や機能発現機構はほとんど同じである。したがって,この高度好熟菌で解明された生命現象のほとんどは,ヒトを含めたあらゆる生物に共通である。
【2.本プロジェクトの進行手順】
1)遺伝子解析(全ゲノムの塩基配列決定)
2)各蛋白賞の量産化
3)各蛋白質の立体構造解析
4)各蛋白質の機能解析
5)機能未知の蛋白賞の機能解析これらを,可能な箇所から同時進行で行う。
1)全ゲノムの塩基配列決定
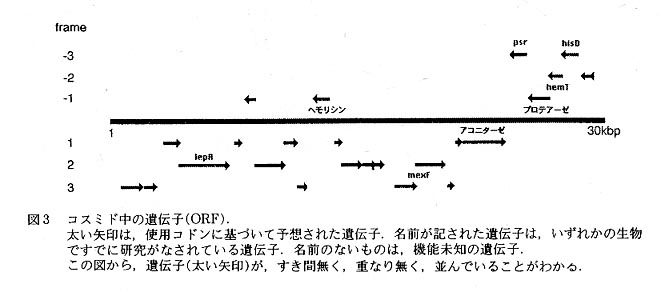
本高度好熱菌DNAはGC含量が70%と高いため,その塩基配列決定は難しいと考えられた時代もあったが,現在は何ら問題を生じていない。逆に,本高度好熱菌のGC含量が高いことを利用すれば,ORFを容易に推定できる(図32))。ゲノム解析は,今年度中に完了する予定である。
2)各蛋白質の宝産化
全蛋白質・約1,500個のうち,これまでに約300個の蛋白質の量産化を手がけている。高度好熱菌蛋白質の量産化には大腸菌を利用する。量産化の目安は,1リットルの培養液から,約100mg(少なくとも10mg程度)の精製蛋白質を得ることである。そのために,T7ファージのプロモーター,または,枯草菌のタンデムプロモーターを持つベクターを用いている。必要に応じて,6×Hisまたは10×HisのHis‐tagを末端に付加させた蛋白質も量産化している。また,以前は難しいと考えられていた膜蛋白質も,in vitro 蛋白質合成系その他の方法で量産化が可能な場合があるので,上記300個の量産化蛋白質の中にl割程度を含めて,量産化を試みている。
これら量産化プラスミドを,近い将来,種々のデータと共に公開する予定である(それ までに入手を希望される場合には,御連絡下さい(kuramitu@bio.sci.osaka-u.ac.jp))。
3)各蛋白質の立体構造解析
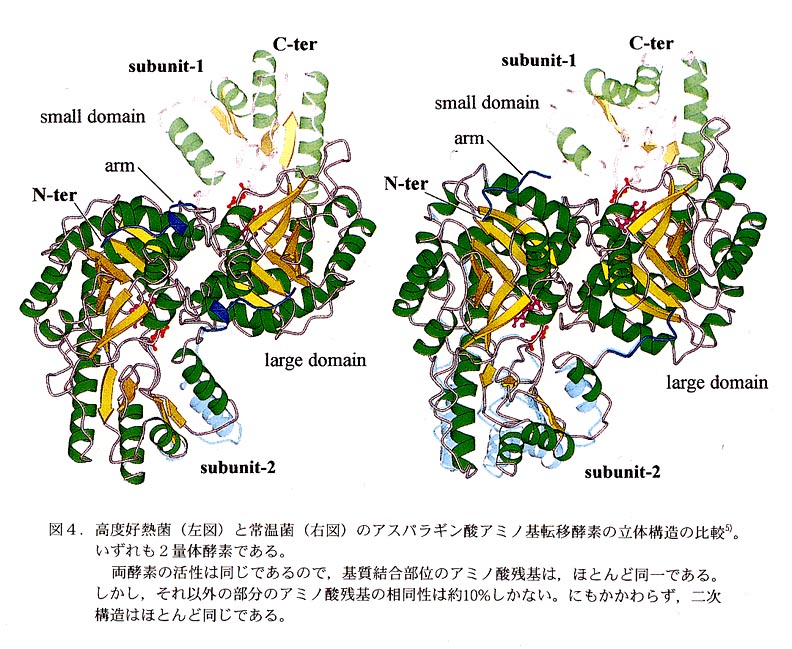
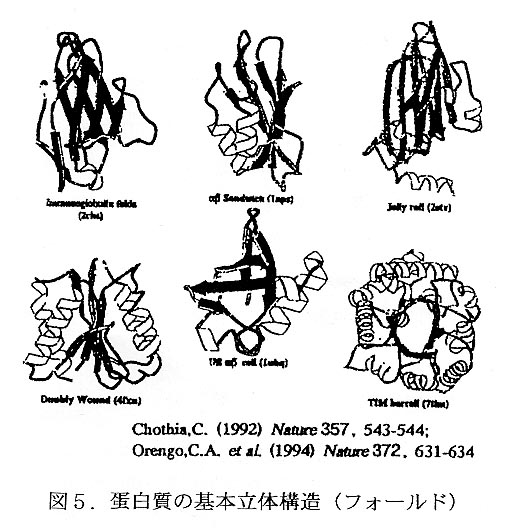
DNAから予想される蛋白質のアミノ酸配列の比較だけではなく,立体構造の比較がいかに重要かを実感したのが,我々のグループで立体構造解析したアミノ基転移酵素の例である(図4)5)。活性部位以外のアミノ酸配列がほとんど異なっていても,ほぼ同じ立体構造をとっていた。
このような立体構造の基本パターン(フオールド)は,約1000種類程度しかないと推定されている6)(図5はその例)。これまでに,約6割がわかっている。外国では,残り4割の新規フォールドをリストアップするために,約10,000個の蛋白質の立体構造を解析するパイロットプロジェクトが始まっている(http://www.nih.gov/nigms/news/meetings/)。
日本では,唯一,「高度好熱菌丸ごと一匹ブロジェクト」の蛋白質の立体構造解析の部分だけが,SPring-8で「ストラクチユローム」研究として始まる。
高度好熱菌T. thermophilus HB8 の蛋白質の立体構造解析からも,相当数の新規フォールドが決定できるであろう。そうなれば,蛋白質の立体構造予測の精度も上がると期待される。
4)各蛋白質の分子機能解析
日本の「高度好熱菌丸ごと一匹プロジェクト」の目標は,まず,高度好熱菌の蛋白質を 集中的に立体構造解析するが,それだけにとどまらず,決定された立体構造に基づいて分子機能解析を行い,最終的には一つの細胞全体の生命現象を原子レベルで総合的に理解しようという(Structural and Functional Genomics の)壮大な計画である。
しかし,この分子機能解析について,分子生物学者・構造生物学者のいずれからも,理解を得ることは難しいのが現状である。
分子機能解析の効率は,立体構造解析が完了すれば飛躍的に上昇する。その分子機能解析の過程の例を図6に示す。
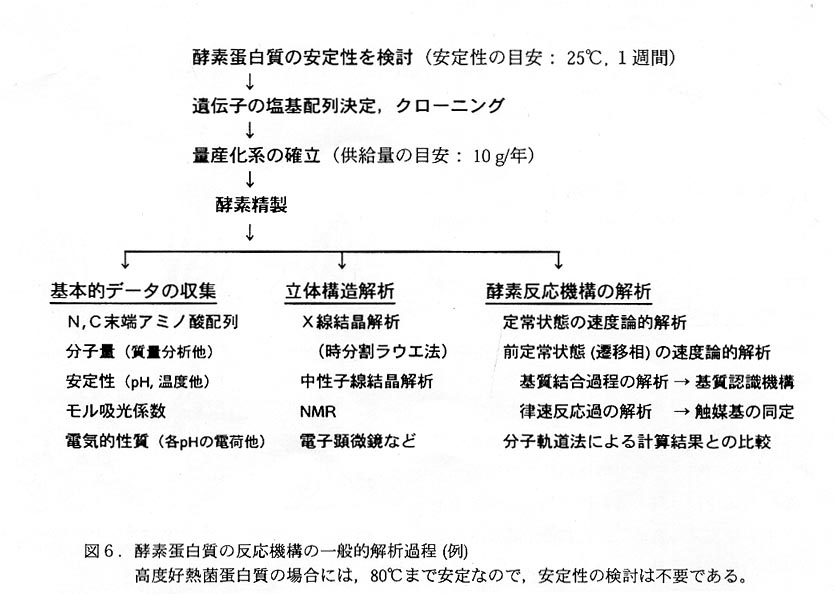
酵素の分子機能を解析する際には,まずX線結晶解析やNMRのような構造生物学的解析 方法で酵素分子の立体構造を決定しておく.一般に酵素反応過程は10個以上の反応素過程からなるので,立体構造から得られる情報は,酵素反応全体を理解するために必要な情報のごく一部である。そこで次に,実際の酵素反応過程を分光学的方法を利用して速度論的に解析し,遷移状態を含めた反応過程の解析を行う。
さらに詳細に,蛋白質中の各アミノ酸残基の役割を調べるために,遺伝子操作でアミノ酸残基を置換して変異型酵素を作製し,その立体構造解析と分子機能解析とを行う。それによって,野生型酵素中におけるそのアミノ酸残基の役割を推定する(図6)。
生物科学においては,新奇な現象は観察されていても測定方法や解析方法が確立していないために統一的理解ができていない諸現象が数多く残されている。それらを理解するためにも,新規な分子機能解析方法の開発を精力的に進める必要がある。
5)機能未知の蛋白質の分子機能解析
チップテクノロジー,温度感受性遺伝子破壊株,蛋白質の二次元電気泳動,立体構造解析,その他現在考え得るあらゆる方法を組み合わせる。
機能未知の立体構造解析を行っても,これまでは業績としてほとんど認められなかった。しかし,たとえ機能未知でも,他の生物に共通のホモログが見い出されるような場合には,その立体構造解析によって,大発見のごく手前まで到達している可能性がある。
幸い,このような機能未知蛋白質の立体構造解析結果を掲載する雑誌が間もなく発刊されるようである。
6)細胞全体の生命現象の理解を目指して
前述のようにして,個々の分子機能を解明した後,複合系生体分子の解析を行い,より高次の生命活動の解析を行う。
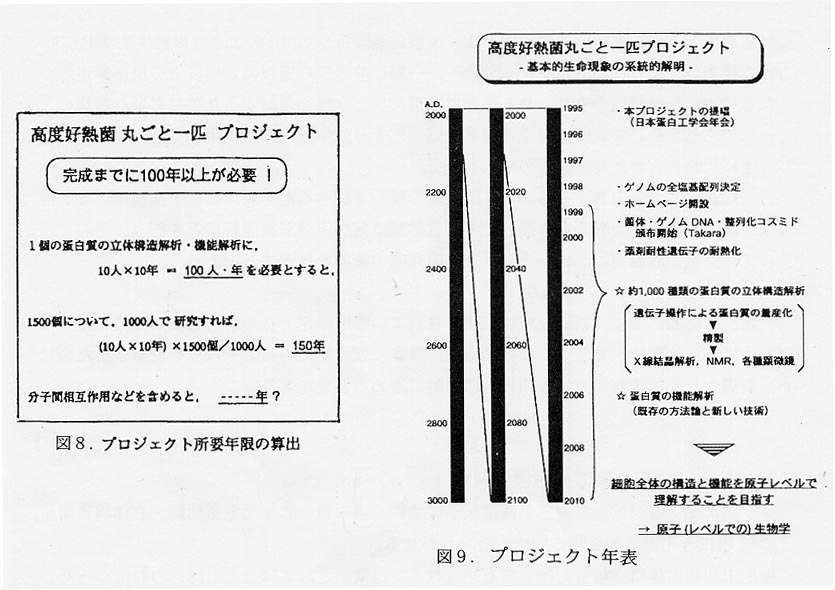
我々の経験によれば,一つの酵素蛋白質について立体構造から分子機能までを徹底して解析しようとすれば,10人の研究者で少なくとも10年問は要する。本高度好熱菌には約1500種類の蛋白質があるので7)(図7),今後解析方法の飛躍的発展が無ければ,一通り研究するだけでも,1000人の研究者で100年以上を要する。さらに,それら複合系の相互作用までも解析しようとすると,気が遠くなるような年月を要する(図8,9)。しかし,その研究過程で得られる成果にはかり知れないものがあることは,容易に想像できる。
このプロジェクト全体は,生命科学の基礎研究分野における壮大である。本気で取り組めば,現在の脳科学や計算機科学以上のプロジェクトとなる。
幸い,高度好熱菌T. thermophilus HB8のゲノム解析・蛋白質の量産化・蛋白質の立体構造解析だけについては,理研の「ストラクチユローム」連携研究を核にして,始めることが可能になった。その立体構造解析結果に基づいて分子機能解析が進み,一つの細胞全体の生命現象の理解へ少しでも近ずくのが夢である。
おわりに
最近では種々の生物の遺伝子が次々と決められ,機能未知遺伝子の機能解明が試みられているが蛋白質の安定性が低かったり,蛋白質の結晶化が難しいために,必ずしも成功していない。また,耐熱生物であっても遺伝子操作系が無いことがある。最初にも述べたように,蛋白質やDNA分子の立体構造に基づいて細胞全体を理解しようとするならば,遺伝子操作系が確立した生物の中で,もっとも耐熱性が高いもの(T. thermophilus HB8)を選ぶのが得策と考えられる。さらにこの高度好熱菌のように小さなゲノムに残された遺伝子は,あらゆる生物に必須で共通な遺伝子を持っている確率が高い。したがって,構造生物学的解析法その他の解析法を駆使してこの高度好熱菌を系統的に研究することは,基本的生命現象を総合的に理解するための近道であると考えられる。
たかが一匹の小さな高度好熱菌だが,そのプロジェクトの壮大さに気付いた時,「このプロジエクトは,少数グループのための研究ではなく,未来の人類のためのボランテイア的基礎研究である。この連携研究の成否は,いかに研究者が協力し合えるかにかかっている。」と思うと同時に,坂部プロジェクトの献身的運営方針を手本にしたいと思っている。
【参考文献】